



生物多样性 ›› 2025, Vol. 33 ›› Issue (9): 25270. DOI: 10.17520/biods.2025270 cstr: 32101.14.biods.2025270
旦增尼玛1,#( ), 孙伟2,#(
), 孙伟2,#( ), 李聪2(
), 李聪2( ), 张纾意2(
), 张纾意2( ), 赵竹楠2(
), 赵竹楠2( ), 许永强1(
), 许永强1( ), 普布卓玛1(
), 普布卓玛1( ), 罗诗琦2(
), 罗诗琦2( ), 达娃1,*(
), 达娃1,*( )(
)( ), 周欣2,*(
), 周欣2,*( )(
)( )
)
收稿日期:2025-07-14
接受日期:2025-08-20
出版日期:2025-09-20
发布日期:2025-10-31
通讯作者:
*E-mail: tsea2@163.com; xinzhoucaddis@icloud.com
作者简介:#共同第一作者
基金资助:
Tenzin Nyima1,#( ), Wei Sun2,#(
), Wei Sun2,#( ), Cong Li2(
), Cong Li2( ), Shuyi Zhang2(
), Shuyi Zhang2( ), Zhunan Zhao2(
), Zhunan Zhao2( ), Yongqiang Xu1(
), Yongqiang Xu1( ), Zhuoma Pubu1(
), Zhuoma Pubu1( ), Shiqi Luo2(
), Shiqi Luo2( ), Wa Da1,*(
), Wa Da1,*( )(
)( ), Xin Zhou2,*(
), Xin Zhou2,*( )(
)( )
)
Received:2025-07-14
Accepted:2025-08-20
Online:2025-09-20
Published:2025-10-31
Contact:
*E-mail: tsea2@163.com; xinzhoucaddis@icloud.com
About author:#Co-first authors
Supported by:摘要:
西藏吉隆沟生物多样性丰富, 其中以传粉昆虫为主要媒介的传粉网络在维护生态系统的稳定性中发挥了重要作用。为了更高效地对当地传粉网络的组成和变化、以及关键传粉昆虫的食性组成等开展监测研究, 本研究拟构建西藏吉隆沟的开花植物叶绿体数据库以用于花粉植物种类的分子鉴定。首先, 我们基于中国数字植物标本馆数据, 整理了吉隆沟6-11月份开花植物名录。通过NCBI公共数据库下载吉隆沟名录物种中已发表的叶绿体基因组数据, 并基于高通量测序数据新组装和注释了9种植物的叶绿体基因组, 其中包括3种在我国仅分布于西藏地区的植物物种: 美丽唐松草(Thalictrum reniforme)、尼泊尔锦鸡儿(Caragana sukiensis)以及耳叶象牙参(Roscoea auriculata) (近危)。随后, 我们对每种植物的64个蛋白质编码基因(protein coding genes, PCGs)进行提取拼接以作为数据库参考序列。阶段性的吉隆沟开花植物叶绿体基因组数据库包含了419个开花植物物种。该数据库的建立将为吉隆沟传粉昆虫食物多样性组成和传粉偏好的分子鉴定提供重要的数据基础。
数据库(集)基本信息简介
数据库(集)基本信息简介
| 数据库(集)名称 | 西藏吉隆沟地区开花植物叶绿体基因组数据集 |
| 作者 | 旦增尼玛, 孙伟, 李聪, 张纾意, 赵竹楠, 许永强, 普布卓玛, 罗诗琦, 达娃, 周欣 |
| 通讯作者 | 达娃(tsea2@163.com); 周欣(xinzhoucaddis@icloud.com) |
| 时间范围 | 2023-2024年 |
| 地理区域 | 西藏日喀则市吉隆县(28°16′-29°18′ N, 80°20′-86°09′ E) |
| 文件大小 | 25.63 MB |
| 数据格式 | *.fasta; *.xlsx; *.txt |
| 数据链接 | https://doi.org/10.57760/sciencedb.j00152.00042 https://www.biodiversity-science.net/fileup/1005-0094/DATA/2025270.zip |
| 数据库(集)组成 | 数据集共包括1个序列数据文件, 1个数据库样本信息文件, 1个吉隆沟所有开花植物信息记录文件, 9种植物叶绿体基因组注释信息文件。 |
旦增尼玛, 孙伟, 李聪, 张纾意, 赵竹楠, 许永强, 普布卓玛, 罗诗琦, 达娃, 周欣 (2025) 西藏吉隆沟地区开花植物叶绿体基因组数据集. 生物多样性, 33, 25270. DOI: 10.17520/biods.2025270.
Tenzin Nyima, Wei Sun, Cong Li, Shuyi Zhang, Zhunan Zhao, Yongqiang Xu, Zhuoma Pubu, Shiqi Luo, Wa Da, Xin Zhou (2025) The chloroplast genome dataset of flowering plants from the Jilong Valley region in Xizang. Biodiversity Science, 33, 25270. DOI: 10.17520/biods.2025270.
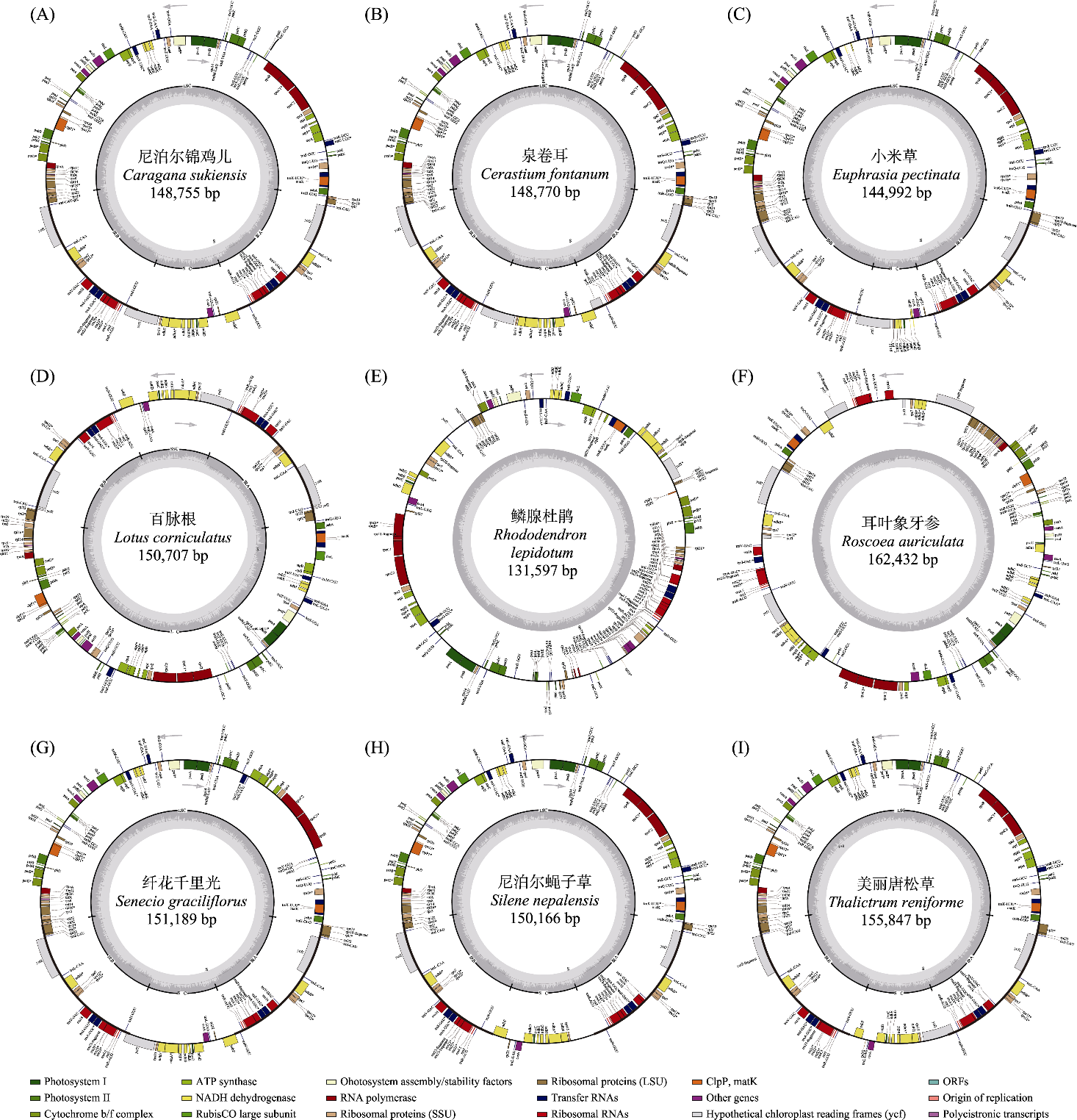
图2 尼泊尔锦鸡儿(A)、泉卷耳(B)、小米草(C)、百脉根(D)、鳞腺杜鹃(E)、耳叶象牙参(F)、纤花千里光(G)、尼泊尔蝇子草(H)和美丽唐松草(I)的叶绿体基因组结构图。外圈显示叶绿体基因组中的基因及其排列和方向; 内圈显示的是不同基因CG含量和调控区。
Fig. 2 Chloroplast genome circular maps of the Caragana sukiensis (A), Cerastium fontanum (B), Euphrasia pectinate (C), Lotus corniculatus (D), Rhododendron lepidotum (E), Roscoea auriculata (F), Senecio graciliflorus (G), Silene nepalensis (H) and Thalictrum reniforme (I). The outer circle of the chloroplast genome circular diagram displays the genes within the chloroplast genome along with their arrangement and orientation; the inner circle shows the CG content of different genes and regulatory regions.
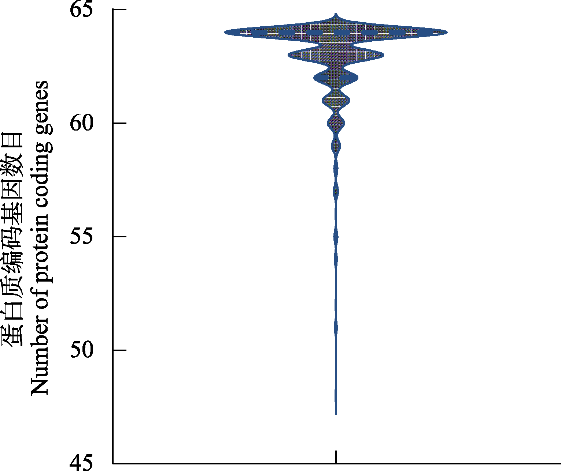
图3 吉隆沟叶绿体基因组数据库中每种植物蛋白质编码基因(PCGs)的数目分布
Fig. 3 The distribution of the number of protein coding genes (PCGs) for each plant in the Jilong Valley chloroplast genome database
| 序号 ID | 基因 Gene | 序号 ID | 基因 Gene | 序号 ID | 基因 Gene | 序号 ID | 基因 Gene |
|---|---|---|---|---|---|---|---|
| 1 | atpA | 17 | petL | 33 | psbL | 49 | rpoC2 |
| 2 | atpB | 18 | petN | 34 | psbM | 50 | rps11 |
| 3 | atpE | 19 | psaA | 35 | psbN | 51 | rps12 |
| 4 | atpF | 20 | psaB | 36 | psbT | 52 | rps14 |
| 5 | atpI | 21 | psaC | 37 | psbZ | 53 | rps15 |
| 6 | ccsA | 22 | psaI | 38 | rbcL | 54 | rps16 |
| 7 | cemA | 23 | psaJ | 39 | rpl14 | 55 | rps18 |
| 8 | clpP | 24 | psbA | 40 | rpl16 | 56 | rps19 |
| 9 | matK | 25 | psbB | 41 | rpl20 | 57 | rps2 |
| 10 | ndhD | 26 | psbC | 42 | rpl22 | 58 | rps3 |
| 11 | ndhF | 27 | psbD | 43 | rpl23 | 59 | rps4 |
| 12 | ndhJ | 28 | psbE | 44 | rpl32 | 60 | rps7 |
| 13 | ndhK | 29 | psbF | 45 | rpl36 | 61 | rps8 |
| 14 | petA | 30 | psbI | 46 | rpoA | 62 | ycf1 |
| 15 | petB | 31 | psbJ | 47 | rpoB | 63 | ycf2 |
| 16 | petD | 32 | psbK | 48 | rpoC1 | 64 | ycf3 |
表1 用于构建数据库的叶绿体蛋白编码基因名单
Table 1 List of chloroplast protein coding genes used for database construction
| 序号 ID | 基因 Gene | 序号 ID | 基因 Gene | 序号 ID | 基因 Gene | 序号 ID | 基因 Gene |
|---|---|---|---|---|---|---|---|
| 1 | atpA | 17 | petL | 33 | psbL | 49 | rpoC2 |
| 2 | atpB | 18 | petN | 34 | psbM | 50 | rps11 |
| 3 | atpE | 19 | psaA | 35 | psbN | 51 | rps12 |
| 4 | atpF | 20 | psaB | 36 | psbT | 52 | rps14 |
| 5 | atpI | 21 | psaC | 37 | psbZ | 53 | rps15 |
| 6 | ccsA | 22 | psaI | 38 | rbcL | 54 | rps16 |
| 7 | cemA | 23 | psaJ | 39 | rpl14 | 55 | rps18 |
| 8 | clpP | 24 | psbA | 40 | rpl16 | 56 | rps19 |
| 9 | matK | 25 | psbB | 41 | rpl20 | 57 | rps2 |
| 10 | ndhD | 26 | psbC | 42 | rpl22 | 58 | rps3 |
| 11 | ndhF | 27 | psbD | 43 | rpl23 | 59 | rps4 |
| 12 | ndhJ | 28 | psbE | 44 | rpl32 | 60 | rps7 |
| 13 | ndhK | 29 | psbF | 45 | rpl36 | 61 | rps8 |
| 14 | petA | 30 | psbI | 46 | rpoA | 62 | ycf1 |
| 15 | petB | 31 | psbJ | 47 | rpoB | 63 | ycf2 |
| 16 | petD | 32 | psbK | 48 | rpoC1 | 64 | ycf3 |
| [1] |
Bagella S, Satta A, Floris I, Caria MC, Rossetti I, Podani J (2013) Effects of plant community composition and flowering phenology on honeybee foraging in Mediterranean sylvo-pastoral systems. Applied Vegetation Science, 16, 689-697.
DOI URL |
| [2] |
Bell KL, de Vere N, Keller A, Richardson RT, Gous A, Burgess KS, Brosi BJ (2016) Pollen DNA barcoding: Current applications and future prospects. Genome, 59, 629-640.
DOI PMID |
| [3] |
Bell KL, Petit RA III, Cutler A, Dobbs EK, MacPherson JM, Read TD, Burgess KS, Brosi BJ (2021) Comparing whole-genome shotgun sequencing and DNA metabarcoding approaches for species identification and quantification of pollen species mixtures. Ecology and Evolution, 11, 16082-16098.
DOI PMID |
| [4] |
Chen SF, Zhou YQ, Chen YR, Gu J (2018) fastp: An ultra-fast all-in-one FASTQ preprocessor. Bioinformatics, 34, 884-890.
DOI PMID |
| [5] |
Corso G, Torres Cruz CP, Pinto MP, de Almeida AM, Lewinsohn TM (2015) Binary versus weighted interaction networks. Ecological Complexity, 23, 68-72.
DOI URL |
| [6] |
Crampton-Platt A, Yu DW, Zhou X, Vogler AP (2016) Mitochondrial metagenomics: Letting the genes out of the bottle. GigaScience, 5, 15.
DOI PMID |
| [7] | Dierckxsens N, Mardulyn P, Smits G (2017) NOVOPlasty: de novo assembly of organelle genomes from whole genome data. Nucleic Acids Research, 45, e18. |
| [8] |
Fang Q, Huang SQ (2012) Progress in pollination networks: Network structure and dynamics. Biodiversity Science, 20, 300-307. (in Chinese with English abstract)
DOI |
|
[方强, 黄双全 (2012) 传粉网络的研究进展: 网络的结构和动态. 生物多样性, 20, 300-307.]
DOI |
|
| [9] | Greiner S, Lehwark P, Bock R (2019) OrganellarGenomeDRAW (OGDRAW) version 1.3.1: Expanded toolkit for the graphical visualization of organellar genomes. Nucleic Acids Research, 47, W59-W64. |
| [10] |
Hua ZY, Tian DM, Jiang C, Song SH, Chen ZY, Zhao YY, Jin Y, Huang LQ, Zhang Z, Yuan Y (2022) Towards comprehensive integration and curation of chloroplast genomes. Plant Biotechnology Journal, 20, 2239-2241.
DOI PMID |
| [11] |
Jin JJ, Yu WB, Yang JB, Song Y, DePamphilis CW, Yi TS, Li DZ (2020) GetOrganelle: A fast and versatile toolkit for accurate de novo assembly of organelle genomes. Genome Biology, 21, 241.
DOI |
| [12] | Kaiser-Bunbury CN, Mougal J, Whittington AE, Valentin T, Gabriel R, Olesen JM, Blüthgen N (2017) Ecosystem restoration strengthens pollination network resilience and function. Nature, 542, 223-227. |
| [13] |
Khansari E, Zarre S, Alizadeh K, Attar F, Aghabeigi F, Salmaki Y (2012) Pollen morphology of Campanula (Campanulaceae) and allied genera in Iran with special focus on its systematic implication. Flora, 207, 203-211.
DOI URL |
| [14] |
Lamb PD, Hunter E, Pinnegar JK, Creer S, Davies RG, Taylor MI (2019) How quantitative is metabarcoding: A meta-analytical approach. Molecular Ecology, 28, 420-430.
DOI PMID |
| [15] |
Lang DD, Tang M, Hu JH, Zhou X (2019) Genome-skimming provides accurate quantification for pollen mixtures. Molecular Ecology Resources, 19, 1433-1446.
DOI PMID |
| [16] |
Li H, Durbin R (2009) Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 25, 1754-1760.
DOI PMID |
| [17] |
Pornon A, Escaravage N, Burrus M, Holota H, Khimoun A, Mariette J, Pellizzari C, Iribar A, Etienne R, Taberlet P, Vidal M, Winterton P, Zinger L, Andalo C (2016) Using metabarcoding to reveal and quantify plant-pollinator interactions. Scientific Reports, 6, 27282.
DOI PMID |
| [18] |
Potts SG, Biesmeijer JC, Kremen C, Neumann P, Schweiger O, Kunin WE (2010) Global pollinator declines: Trends, impacts and drivers. Trends in Ecology & Evolution, 25, 345-353.
DOI URL |
| [19] |
Potts SG, Imperatriz-Fonseca V, Ngo HT, Aizen MA, Biesmeijer JC, Breeze TD, Dicks LV, Garibaldi LA, Hill R, Settele J, Vanbergen AJ (2016) Safeguarding pollinators and their values to human well-being. Nature, 540, 220-229.
DOI |
| [20] |
Taberlet P, Coissac E, Pompanon F, Brochmann C, Willerslev E (2012) Towards next-generation biodiversity assessment using DNA metabarcoding. Molecular Ecology, 21, 2045-2050.
DOI PMID |
| [21] | Tillich M, Lehwark P, Pellizzer T, Ulbricht-Jones ES, Fischer A, Bock R, Greiner S (2017) GeSeq—Versatile and accurate annotation of organelle genomes. Nucleic Acids Research, 45, W6-W11. |
| [1] | 李慧霞, 李玉, 宁馨, 李晓晨, 王天瑞, 宋以刚, 戴锡玲, 郑斯斯, 钟鑫. 基于叶绿体基因组的江南牡丹草遗传多样性与遗传结构[J]. 生物多样性, 2025, 33(8): 25149-. |
| [2] | 孙婷婷, 张彦文. 花粉二型性研究进展[J]. 生物多样性, 2025, 33(7): 25031-. |
| [3] | 廖雅晴, 黄泽锋, 王晓云, 张礼标, 吴毅, 余文华. 广东省翼手目物种名录更新及分子条形码数据库[J]. 生物多样性, 2025, 33(4): 24584-. |
| [4] | 巴苏艳, 赵春艳, 刘媛, 方强. 通过虫体花粉识别构建植物‒传粉者网络: 人工模型与AI模型高度一致[J]. 生物多样性, 2024, 32(6): 24088-. |
| [5] | 何凯莹, 徐心慧, 张承云, 郝泽周, 肖治术, 郭莹莹. 生物声学数据档案的管理标准及管理技术进展[J]. 生物多样性, 2024, 32(10): 24266-. |
| [6] | 吕晓琴, 李杨, 王顺雨, 姚仁秀, 王晓月. 中甸乌头总状花序不同位置花粉和花蜜的化学性状没有显著差异[J]. 生物多样性, 2024, 32(1): 23371-. |
| [7] | 张飞飞, 杨天凤, 陈莉荣, 刘冬梅, 杨柳园, 杨杜宇, 鞠鹏, 陆露. 被子植物花粉颜色多样性及应用研究进展[J]. 生物多样性, 2024, 32(1): 23346-. |
| [8] | 邢超, 林依, 周智强, 赵联军, 蒋仕伟, 林蓁蓁, 徐基良, 詹祥江. 基于DNA条形码技术构建王朗国家级自然保护区陆生脊椎动物遗传资源数据库及物种鉴定[J]. 生物多样性, 2023, 31(7): 22661-. |
| [9] | 吴帆, 刘深云, 江虎强, 王茜, 陈开威, 李红亮. 中华蜜蜂和意大利蜜蜂秋冬期传粉植物多样性比较[J]. 生物多样性, 2023, 31(5): 22528-. |
| [10] | 丁洪波, 周仕顺, 李剑武, 申健勇, 马兴达, 黄健, 宋钰, 文雪梅, 雷鸣, 土艳丽, 星耀武, 谭运洪. 中国西藏种子植物区系新资料[J]. 生物多样性, 2022, 30(8): 22085-. |
| [11] | 胡德美, 姚仁秀, 陈燕, 游贤松, 王顺雨, 汤晓辛, 王晓月. 青篱柴通过促进亲和花粉生长而提高传粉精确性[J]. 生物多样性, 2021, 29(7): 887-896. |
| [12] | 都业勤, 张迪, 王赛, 王磊, 闫兴富, 唐占辉. 湿地植物大花百合种群的性系统特征[J]. 生物多样性, 2021, 29(10): 1321-1335. |
| [13] | 刘雪华, 张语克, 赵翔宇, 何祥博, 蔡琼, 朱云, 何百锁, 酒强. 秦岭中段野生动物多样性的红外相机监测数据库平台介绍[J]. 生物多样性, 2020, 28(9): 1075-1080. |
| [14] | 万雅琼, 李佳琦, 杨兴文, 李晟, 徐海根. 基于红外相机的中国哺乳动物多样性观测网络建设进展[J]. 生物多样性, 2020, 28(9): 1115-1124. |
| [15] | 王科,赵明君,苏锦河,杨柳,邓红,王永会,吴海军,李熠,吴红梅,卫晓丹,魏铁铮,蔡磊,姚一建. 中国菌物名录数据库在大型真菌红色名录编制中的作用[J]. 生物多样性, 2020, 28(1): 74-98. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
备案号:京ICP备16067583号-7
Copyright © 2022 版权所有 《生物多样性》编辑部
地址: 北京香山南辛村20号, 邮编:100093
电话: 010-62836137, 62836665 E-mail: biodiversity@ibcas.ac.cn