



生物多样性 ›› 2025, Vol. 33 ›› Issue (1): 24403. DOI: 10.17520/biods.2024403 cstr: 32101.14.biods.2024403
贺加贝1( ), 柯可1, 孙海明2, 胡丽萍1, 赵晓伟1, 王文豪1, 赵强1,*(
), 柯可1, 孙海明2, 胡丽萍1, 赵晓伟1, 王文豪1, 赵强1,*( )
)
收稿日期:2024-09-08
接受日期:2025-01-17
出版日期:2025-01-20
发布日期:2025-01-27
通讯作者:
* E-mail: 基金资助:
Jiabei He1( ), Ke Ke1, Haiming Sun2, Liping Hu1, Xiaowei Zhao1, Wenhao Wang1, Qiang Zhao1,*(
), Ke Ke1, Haiming Sun2, Liping Hu1, Xiaowei Zhao1, Wenhao Wang1, Qiang Zhao1,*( )
)
Received:2024-09-08
Accepted:2025-01-17
Online:2025-01-20
Published:2025-01-27
Contact:
* E-mail: Supported by:摘要:
底栖动物是海洋生态系统的重要组成部分, 在调控海洋生态系统的物质循环和能量流动过程中扮演了关键角色。腹足类动物通过捕食与被捕食行为调控着底栖生态系统的稳定性, 研究腹足类生物的食性有助于我们理解这一调控过程。香螺(Neptunea cumingii)是我国北方海域一种重要的腹足类动物, 具备极高的生态价值和经济价值。但是, 我们对其食性组成及生态功能的了解并不透彻。因此, 有必要探明自然条件下香螺现场食物组成, 提升对腹足类动物在我国北方海域底栖生态系统中所起调控作用的认识。本研究借助4份野生香螺胃含物样品, 以18S rDNA V4区和V9区为标靶, 利用DNA宏条形码技术对其胃含物真核生物进行分析。结果显示, 在4个样品中, 18S rDNA V4区和V9区共分别获得265,161条和221,998条高质量序列, 分别占各自原始序列的93.16%和86.54%, 分别注释到141个和490个OTUs; 虽然18S rDNA V4区获得的优质序列数及占比均更高, 但其注释到的物种数比18S rDNA V9区偏少。两个可变区所有OTUs分属17个门类, 包括动物界10门、真菌界5门、植物界1门, 以及SAR超类群, 包括不等鞭毛生物(Stramenopiles)、囊泡虫(Alveolates)和有孔虫(Rhizaria); 在纲水平上, 腹足纲、辅鳍鱼纲、吸虫纲和色矛纲相对丰度在两个可变区中均排名前十; 在OTU水平上, 两个可变区中3个以上样品中均有检出的物种仅分别占5.67%和8.08%, 且其共同属于软体动物门、脊椎动物亚门、子囊菌门及SAR超类群。总体上, 18S rDNA V4区和V9区两段DNA条形码分析结果显示, 香螺胃含物中真核生物种类丰富, 包括动物(如环节动物、节肢动物、软体动物)、真菌、植物和原生生物, 其中最丰富的类群是腹足类、鱼类、吸虫和真菌。在多个样本中只检测到一小部分共有OTUs, 这表明不同香螺摄食种类多变。结果表明, 香螺现场食性是一种机会主义捕食者, 动物尸体及海底沉积物可能是香螺自然状态下主要食物来源, 但同时其具备一定的清理附着生物潜能及植食性能力, 饵料可驯化性较强。其饵料组成受生存微环境影响较大, 具备一定的饵料驯化潜力。研究结果为深入了解香螺在海洋生态系统中的作用提供了数据支持, 并为香螺人工养殖饵料配比研究提供了新见解。
贺加贝, 柯可, 孙海明, 胡丽萍, 赵晓伟, 王文豪, 赵强 (2025) 基于DNA宏条形码技术分析香螺食性. 生物多样性, 33, 24403. DOI: 10.17520/biods.2024403.
Jiabei He, Ke Ke, Haiming Sun, Liping Hu, Xiaowei Zhao, Wenhao Wang, Qiang Zhao (2025) Diet analysis of Neptunea cumingii using metabarcoding. Biodiversity Science, 33, 24403. DOI: 10.17520/biods.2024403.
| 引物 Primer | 引物序列 Primer sequence | 预期长度 Fragment length | 参考文献 Reference | |
|---|---|---|---|---|
| 18S rDNA V4 | 3NDF | 5′-GGCAAGTCTGGTGCCAG-3′ | 450 bp | Bråte et al, |
| V4-euk-R2R | 5′-ACGGTATCTRATCRTCTTCG-3′ | |||
| 18S rDNA V9 | Euk1391f | 5′-GTACACACCGCCCGTC-3′ | 200‒300 bp | Amaral-Zettler et al, |
| EukBr | 5′-TGATCCTTCTGCAGGTTCACCTAC-3′ |
表1 基于18S rDNA不同可变区对香螺胃含物种类鉴定使用的PCR引物
Table 1 PCR primers for species identification for stomach contents of Neptunea cumingii based on different hypervariable regions of 18S rDNA
| 引物 Primer | 引物序列 Primer sequence | 预期长度 Fragment length | 参考文献 Reference | |
|---|---|---|---|---|
| 18S rDNA V4 | 3NDF | 5′-GGCAAGTCTGGTGCCAG-3′ | 450 bp | Bråte et al, |
| V4-euk-R2R | 5′-ACGGTATCTRATCRTCTTCG-3′ | |||
| 18S rDNA V9 | Euk1391f | 5′-GTACACACCGCCCGTC-3′ | 200‒300 bp | Amaral-Zettler et al, |
| EukBr | 5′-TGATCCTTCTGCAGGTTCACCTAC-3′ |
| 原始序列 Paired-end reads | 高质量序列 High quality reads | 序列平均长度 Average length of reads | OTU数 No. of OTU | |
|---|---|---|---|---|
| 18S rDNA V4 | 284,619 | 265,161 | 429 | 141 |
| 18S rDNA V9 | 256,536 | 221,998 | 124 | 490 |
表2 香螺胃含物中基于18S rDNA不同可变区获得的原始序列数、高质量序列数和操作分类单元数(OTUs)
Table 2 The raw reads, high quality reads and operational taxonomic units (OTUs) in stomach contents of Neptunea cumingii based on different hypervariable regions of 18S rDNA
| 原始序列 Paired-end reads | 高质量序列 High quality reads | 序列平均长度 Average length of reads | OTU数 No. of OTU | |
|---|---|---|---|---|
| 18S rDNA V4 | 284,619 | 265,161 | 429 | 141 |
| 18S rDNA V9 | 256,536 | 221,998 | 124 | 490 |
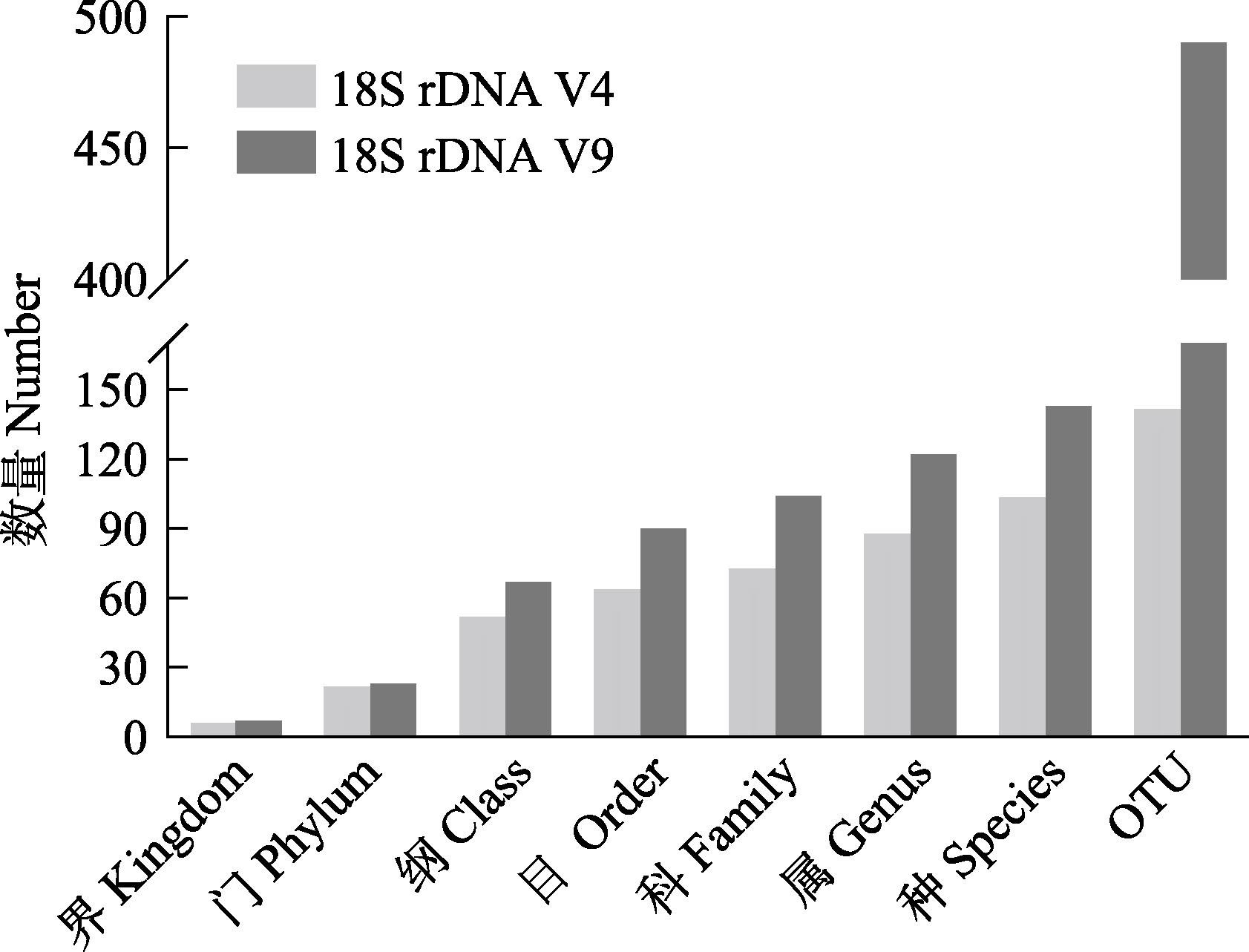
图1 基于不同可变区序列所获香螺胃含物分类信息
Fig. 1 Classification information of stomach contents in Neptunea cumingii obtained from different hypervariable regions. OTU, Operational taxonomic unit.
| 分类 Classification | 细化分类单元 Classification in details |
|---|---|
| 环节动物门 Annelida | 单向蚓目 Haplotaxida |
| 节肢动物门 Arthropoda | 猛水蚤目 Harpacticoida 尾肢目 Podocopida 软甲纲 Malacostraca 膜翅目 Hymenoptera 鳃足纲 Diplostraca 蛛形纲 Arachnida |
| 软体动物门 Mollusca | 牡蛎目 Ostreoida 新腹足目 Caenogastropoda 枪形目 Teuthida 头足纲 Cephalopoda |
| 扁形动物门 Platyhelminthes | 后睾目 Opisthorchiida 斜睾目 Plagiorchiida 独孤科 Azygiida |
| 脊椎动物亚门 Vertebrata | 哺乳纲 Mammalia 辅鳍鱼纲 Actinopterygii |
| 刺胞动物门 Cnidaria | 多壳目 Multivalvulida |
| 眼虫门 Euglenozoa | 异线虫目 Heteronematina 动质体纲 Kinetoplastea 副牙形目 Parabodonida |
| 线虫动物门 Nematozoa | 蛔目 Ascaridida 单宫目 Monhysterida 杆形目 Rhabditida |
| 棘皮动物门 Echinodermata | 海星纲 Asteroidea |
| 苔藓虫门 Bryozoa | 唇口目 Cheilostomatida 栉口目 Ctenostomatida |
| Aphelidea | 未鉴定 Norank |
| 子囊菌门 Ascomycota | 枝孢菌科 Cladosporiaceae 曲霉科 Aspergillaceae 梅奇酵母科 Metschnikowiaceae 毕赤酵母科 Pichiaceae 汉斯德巴酵母科 Debaryomycetaceae 双足囊菌科 Dipodascaceae 亚隔孢壳科 Didymellaceae 酵母科 Saccharomycetaceae 发菌科 Trichocomaceae Plectosphaerellaceae |
| 担子菌门 Basidiomycota | 线黑粉菌科 Filobasidiaceae 马拉色菌科 Malasseziaceae 微球黑粉菌纲 Microbotryomycetes 短臂担科 Brachybasidiaceae 伞菌目 Agaricales 毛孢子菌科 Trichosporonaceae 黑粉菌科 Ustilaginaceae 多孔菌目 Polyporales |
| 壶菌门 Chytridiomycota | 未鉴定 Norank |
| 隐霉菌门 Cryptomycota | 未鉴定 Norank |
| 芦苇藻门 Phragmoplastophyta | 有胚植物纲 Embryophyta |
| SAR超类群 SAR | 网黏菌纲 Labyrinthulomycetes 脆杆菌目 Fragilariales 水云目 Ectocarpales 间藻纲 Mediophyceae 卡盾藻纲 Chattonellales Archigregarinorida Thecofilosea 海洋原生藻 MAST-12A 叶咽纲 Phyllopharyngea Eugregarinorida 横列甲藻纲 Dinophyceae Gregarinasina 植黏菌纲 Phytomyxea 多甲藻目 Peridiniales 直链藻属 Melosirids Suessiaceae |
表3 基于饵料种类分析结果
Table 3 Results of food types analysis based on OTUs
| 分类 Classification | 细化分类单元 Classification in details |
|---|---|
| 环节动物门 Annelida | 单向蚓目 Haplotaxida |
| 节肢动物门 Arthropoda | 猛水蚤目 Harpacticoida 尾肢目 Podocopida 软甲纲 Malacostraca 膜翅目 Hymenoptera 鳃足纲 Diplostraca 蛛形纲 Arachnida |
| 软体动物门 Mollusca | 牡蛎目 Ostreoida 新腹足目 Caenogastropoda 枪形目 Teuthida 头足纲 Cephalopoda |
| 扁形动物门 Platyhelminthes | 后睾目 Opisthorchiida 斜睾目 Plagiorchiida 独孤科 Azygiida |
| 脊椎动物亚门 Vertebrata | 哺乳纲 Mammalia 辅鳍鱼纲 Actinopterygii |
| 刺胞动物门 Cnidaria | 多壳目 Multivalvulida |
| 眼虫门 Euglenozoa | 异线虫目 Heteronematina 动质体纲 Kinetoplastea 副牙形目 Parabodonida |
| 线虫动物门 Nematozoa | 蛔目 Ascaridida 单宫目 Monhysterida 杆形目 Rhabditida |
| 棘皮动物门 Echinodermata | 海星纲 Asteroidea |
| 苔藓虫门 Bryozoa | 唇口目 Cheilostomatida 栉口目 Ctenostomatida |
| Aphelidea | 未鉴定 Norank |
| 子囊菌门 Ascomycota | 枝孢菌科 Cladosporiaceae 曲霉科 Aspergillaceae 梅奇酵母科 Metschnikowiaceae 毕赤酵母科 Pichiaceae 汉斯德巴酵母科 Debaryomycetaceae 双足囊菌科 Dipodascaceae 亚隔孢壳科 Didymellaceae 酵母科 Saccharomycetaceae 发菌科 Trichocomaceae Plectosphaerellaceae |
| 担子菌门 Basidiomycota | 线黑粉菌科 Filobasidiaceae 马拉色菌科 Malasseziaceae 微球黑粉菌纲 Microbotryomycetes 短臂担科 Brachybasidiaceae 伞菌目 Agaricales 毛孢子菌科 Trichosporonaceae 黑粉菌科 Ustilaginaceae 多孔菌目 Polyporales |
| 壶菌门 Chytridiomycota | 未鉴定 Norank |
| 隐霉菌门 Cryptomycota | 未鉴定 Norank |
| 芦苇藻门 Phragmoplastophyta | 有胚植物纲 Embryophyta |
| SAR超类群 SAR | 网黏菌纲 Labyrinthulomycetes 脆杆菌目 Fragilariales 水云目 Ectocarpales 间藻纲 Mediophyceae 卡盾藻纲 Chattonellales Archigregarinorida Thecofilosea 海洋原生藻 MAST-12A 叶咽纲 Phyllopharyngea Eugregarinorida 横列甲藻纲 Dinophyceae Gregarinasina 植黏菌纲 Phytomyxea 多甲藻目 Peridiniales 直链藻属 Melosirids Suessiaceae |
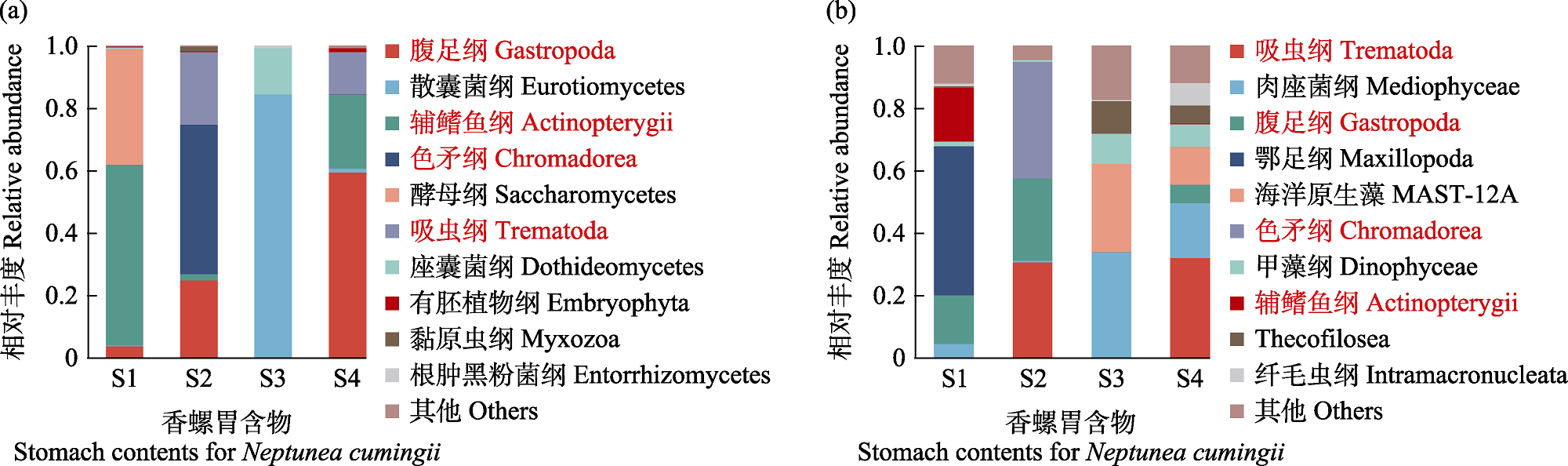
图2 香螺胃含物纲水平相对丰度组成(S1‒4代表不同样品)。(a) 18S rDNA V4区; (b) 18S rDNA V9区。图例中红色字体为18S rDNA V4和V9区共同检出类别。
Fig. 2 Relative abundance of stomach contents for Neptunea cumingii in the class level (S1‒4 represents different samples). (a) 18S rDNA V4 hypervariable region; (b) 18S rDNA V9 hypervariable region. The red font in the legend indicates the taxonomic categories identified by both the 18S rDNA V4 and V9 regions.
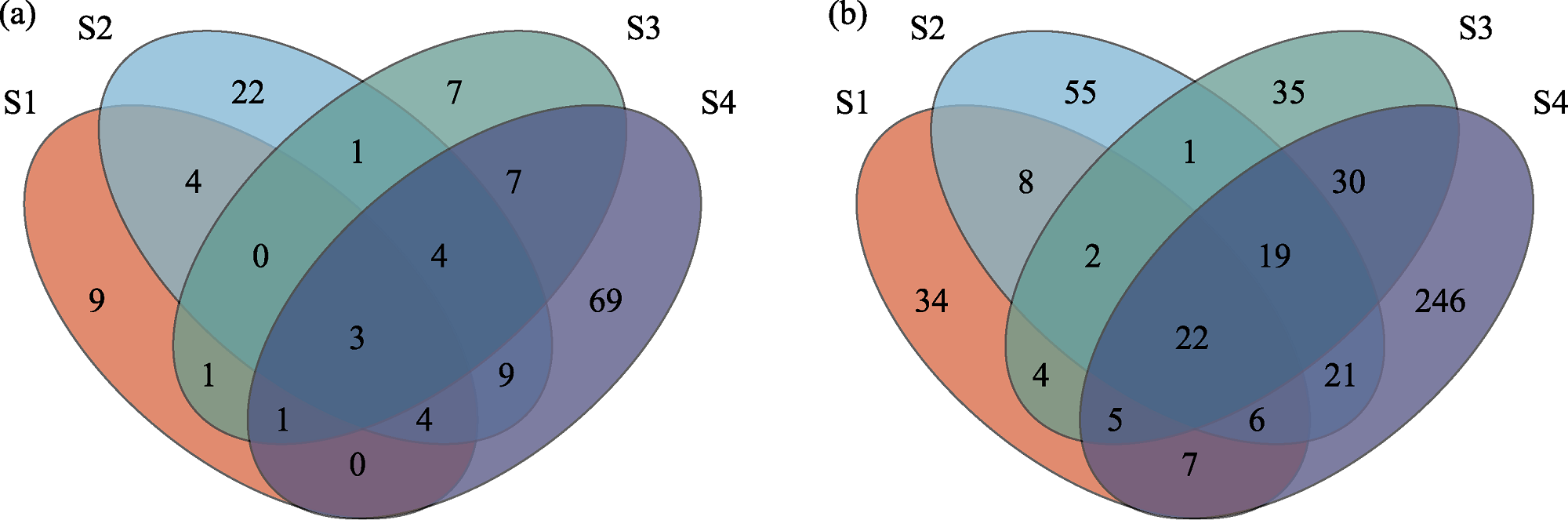
图3 香螺胃含物不同样品中OTUs分布韦恩图(S1‒4代表不同样品)。(a) 18S rDNA V4区; (b) 18S rDNA V9区。
Fig. 3 Venn diagram showing the unique and shared OTUs in samples (S1-4 represents different samples). (a) 18S rDNA V4 hypervariable region; (b) 18S rDNA V9 hypervariable region.
| [1] | Amaral-Zettler LA, McCliment EA, Ducklow HW, Huse SM (2009) A method for studying protistan diversity using massively parallel sequencing of V9 hypervariable regions of small-subunit ribosomal RNA genes. PLoS ONE, 4, e6372. |
| [2] | Bråte J, Logares R, Berney C, Ree DK, Klaveness D, Jakobsen KS, Shalchian-Tabrizi K (2010) Freshwater Perkinsea and marine-freshwater colonizations revealed by pyrosequencing and phylogeny of environmental rDNA. The ISME Journal, 4, 1144-1153. |
| [3] | Cai DL, Hong XG, Mao XH, Zhang SF, Han YB, Gao SL (2001) Preliminary studies on trophic structure of tidal zone in the Laoshan Bay by using carbon stable isotopes. Acta Oceanologica Sinica, 23(4), 41-47. (in Chinese with English abstract) |
| [蔡德陵, 洪旭光, 毛兴华, 张淑芳, 韩贻兵, 高素兰 (2001) 崂山湾潮间带食物网结构的碳稳定同位素初步研究. 海洋学报, 23(4), 41-47.] | |
| [4] | Cai WJ, Zhang CF, Li KM, Hao JH, Qin MX, Guan HT, Gao Q, Liu D, Nie MM, Jia JM, Qi DL (2023) Intestinal microstructure and dietary analysis based on macrobarcode of Gymnocypris przewalskii. Acta Hydrobiologica Sinica, 47, 1954-1964. (in Chinese with English abstract) |
| [蔡伟杰, 张存芳, 李柯懋, 郝佳慧, 覃敏欣, 关弘弢, 高强, 刘丹, 聂苗苗, 贾军梅, 祁得林 (2023) 青海湖裸鲤肠道显微结构及基于宏条形码的食性分析. 水生生物学报, 47, 1954-1964.] | |
| [5] |
Cong JY, Li XZ, Xu Y (2024) Application of species distribution models in predicting the distribution of marine macrobenthos. Chinese Journal of Applied Ecology, 35, 2392-2400. (in Chinese with English abstract)
DOI |
|
[丛佳仪, 李新正, 徐勇 (2024) 物种分布模型在海洋大型底栖动物分布预测中的应用. 应用生态学报, 35, 2392-2400.]
DOI |
|
| [6] |
Edgar RC (2013) UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 10, 996-998.
DOI PMID |
| [7] | Gao XD, Chen XJ, Li YK (2018) A review on the methods used in aquatic food web research: Development and applications. Journal of Fishery Sciences of China, 25, 1347-1360. (in Chinese with English abstract) |
| [高小迪, 陈新军, 李云凯 (2018) 水生食物网研究方法的发展和应用. 中国水产科学, 25, 1347-1360.] | |
| [8] | Guo YH, Li WJ, Xuan XZ, Li Y (2023) Research progress on application of yeast in culture of Stichopus japonicus. Feed Research, 46(20), 173-177. (in Chinese with English abstract) |
| [郭益红, 李文嘉, 宣雄智, 李烨 (2023) 酵母在刺参养殖中的应用研究进展. 饲料研究, 46(20), 173-177.] | |
| [9] | Hadziavdic K, Lekang K, Lanzen A, Jonassen I, Thompson EM, Troedsson C (2014) Characterization of the 18S rRNA gene for designing universal eukaryote specific primers. PLoS ONE, 9, e87624. |
| [10] | Han WN, Li Z, Pang M, Gong YC, Wu ZX, Zhang ZH, Kang LN, Zhang XL, Wang ZL, Qu P (2024) Differences of carbon and nitrogen stable isotope of Rapana venosa between typical bays and offshore islands. Acta Ecologica Sinica, 44, 4244-4253. (in Chinese with English abstract) |
| [韩温诺, 李曌, 庞敏, 宫于琛, 吴忠迅, 张朝晖, 康丽娜, 张学雷, 王宗灵, 屈佩 (2024) 典型海湾与海岛近海脉红螺碳、氮稳定同位素特征差异. 生态学报, 44, 4244-4253.] | |
| [11] | He JB, Wang H, Zhao YN, Li F, Yang LF, Zhao Q (2024a) Histological observation of reproductive system and reproductive cell characteristics of limpet (Neptunea cumingii) under artificial culture conditions. Journal of Dalian Ocean University, 39, 402-409. (in Chinese with English abstract) |
| [贺加贝, 王涵, 赵延宁, 李峰, 杨陆飞, 赵强 (2024a) 人工养殖条件下香螺生殖系统组织学观察及生殖细胞特征. 大连海洋大学学报, 39, 402-409.] | |
| [12] | He JB, Zhao XW, Ji SL, Wang H, Zhao Q (2024b) Study on feeding behavior of Neptunea arthritica cumingii under artificial culture conditions. Marine Sciences, 48(6), 43-49. (in Chinese with English abstract) |
| [贺加贝, 赵晓伟, 汲生磊, 王涵, 赵强 (2024b) 人工养殖条件下香螺摄食行为研究. 海洋科学, 48(6), 43-49.] | |
| [13] | He JB, Zhao XW, Yang LF, Ji SL, Zhao YX, Zhao Q (2024c) Effects of integrated aquaculture of bivalves and limpet Neptunea cumingii on predation, growth and survival of cultured organisms. Journal of Dalian Ocean University, 39, 788-796. (in Chinese with English abstract) |
| [贺加贝, 赵晓伟, 杨陆飞, 汲生磊, 赵运星, 赵强 (2024c) 贝螺综合养殖模式对养殖对象摄食、生长及存活的影响. 大连海洋大学学报, 39, 788-796.] | |
| [14] | Hebert PDN, Cywinska A, Ball SL, Dewaard JR (2003) Biological identifications through DNA barcodes. Proceedings of the Royal Society of London Series B: Biological Sciences, 270, 313-321. |
| [15] | Hidalgo MC, Urea E, Sanz A (1999) Comparative study of digestive enzymes in fish with different nutritional habits: Proteolytic and amylase activities. Aquaculture, 170, 267-283. |
| [16] | Ji DP (2014) Biological Study on Fishery Resources of Spotted Head Fish and Six-line Fish in Rongcheng Lidao. PhD dissertation, Ocean University of China, Qingdao, Shandong. (in Chinese with English abstract) |
| [纪东平 (2014) 荣成俚岛斑头鱼和大泷六线鱼的渔业资源生物学研究. 博士学位论文, 中国海洋大学, 山东青岛.] | |
| [17] | Jia N, Zhou TC, Hu SM, Zhang C, Huang H, Liu S (2024) Difference in the feeding contents of three hermit crabs in the coral reefs of the Nansha Islands, South China Sea. Journal of Tropical Oceanography, 43(3), 109-121. (in Chinese with English abstract) |
| [贾男, 周天成, 胡思敏, 张琛, 黄晖, 刘胜 (2024) 南沙群岛海域珊瑚礁区三种寄居蟹的摄食差异比较. 热带海洋学报, 43(3), 109-121.] | |
| [18] | Li FX, Du MR, Gao YP, Wang JW, Zhang YT, Zhang ZX, Jiang ZJ (2021) Analysis of food sources of Crassostrea gigas using high-throughput sequencing techniques. Progress in Fishery Sciences, 42(5), 86-96. (in Chinese with English abstract) |
| [李凤雪, 杜美荣, 高亚平, 王军威, 张义涛, 张志新, 蒋增杰 (2021) 长牡蛎食物组成的高通量测序分析. 渔业科学进展, 42(5), 86-96.] | |
| [19] | Li HY, Wang YN, Meng L, Li JZ, Dong SY, Li YR, Guo YJ, Zhang T (2023) Effects of feeding preferences and temperature on Neptunea arthritica cumingii. Marine Sciences, 47(8), 17-22. (in Chinese with English abstract) |
| [李华煜, 王元宁, 孟良, 李金泽, 董诗雨, 李永仁, 郭永军, 张涛 (2023) 饵料、温度对香螺摄食的影响. 海洋科学, 47(8), 17-22.] | |
| [20] | Li JY, Leng Y, Pan YL, Xu ZD, Bao MM, Sha JJ (2019) An ecological research on marine fouling organisms in Shidao Island areas. Transactions of Oceanology and Limnology, 41(6), 139-146. (in Chinese with English abstract) |
| [李继业, 冷宇, 潘玉龙, 徐兆东, 鲍萌萌, 沙婧婧 (2019) 石岛海域污损生物生态学研究. 海洋湖沼通报, 41(6), 139-146.] | |
| [21] | Li Q, Ran K, Kong LF (2021) DNA barcoding and its application in marine shellfish germplasm resources conservation. Periodical of Ocean University of China, 51(5), 18-23. (in Chinese with English abstract) |
| [李琪, 冉轲, 孔令锋 (2021) DNA条形码技术及其在海洋贝类种质资源保护中的应用. 中国海洋大学学报(自然科学版), 51(5), 18-23.] | |
| [22] | Li QY, Chen F, Zhang HL, Qian WG, Jiang RJ (2024) Community structure and distribution characteristics of shellfish in the coastal waters of Zhejiang. Journal of Shanghai Ocean University, 33, 1223-1237. (in Chinese with English abstract) |
| [李庆英, 陈峰, 张洪亮, 钱卫国, 蒋日进 (2024) 浙江近海大型底栖贝类群落结构及其分布特征. 上海海洋大学学报, 33, 1223-1237.] | |
| [23] | Li YL, Chen BL, Bao XB, Zhou ZC, Liu WD, Li YF (2023) Preliminary dietary analysis of Hyporhamphus sajori juveniles based on DNA metabarcoding. Journal of Fishery Sciences of China, 30, 393-405. (in Chinese with English abstract) |
| [李玉龙, 陈百灵, 鲍相渤, 周遵春, 刘卫东, 李云峰 (2023) 基于DNA宏条形码技术的沙氏下鱵幼鱼食性分析. 中国水产科学, 30, 393-405.] | |
| [24] | Stackebrandt E, Goebel BM (1994) Taxonomic note: A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology. International Journal of Systematic and Evolutionary Microbiology, 44, 846-849. |
| [25] | Wang L, Du SC, Yang TY, Chang YQ (2017) Using stable isotopes to evaluate food web structure in Dalian coastal water. Chinese Journal of Ecology, 36, 1452-1457. (in Chinese with English abstract) |
| [王荦, 杜双成, 杨婷越, 常亚青 (2017) 应用稳定同位素技术评价大连近岸海域食物网营养结构. 生态学杂志, 36, 1452-1457.] | |
| [26] | Wang SJ, Ding YH, Jiang MW, Jiang XM, Han QX (2018) Effects of starvation time on growth, biochemical composition, enzyme activities and antioxidant indexes of Hemifusus tuba Gmelin. Chinese Journal of Animal Nutrition, 30, 138-146. (in Chinese with English abstract) |
| [王双健, 丁玉惠, 江茂旺, 蒋霞敏, 韩庆喜 (2018) 饥饿时间对管角螺生长、生化组成、消化酶活性和抗氧化指标的影响. 动物营养学报, 30, 138-146.] | |
| [27] | Wang YP, Li JQ, Xue SY, Chang LR, Lu LF, Zhang YT, Mao YZ (2023) Effects of three kinds of snails on the control of oysters attached to the shell of cultured abalone. Journal of Fishery Sciences of China, 30, 344-351. (in Chinese with English abstract) |
| [王英朴, 李加琦, 薛素燕, 常丽荣, 卢龙飞, 张义涛, 毛玉泽 (2023) 3种螺对养殖鲍外壳附着牡蛎的防除作用. 中国水产科学, 30, 344-351.] | |
| [28] | Xi XQ, Bao BL, Zhang SY (2015) Application of DNA barcoding in species analysis of fish stomach content. Journal of Shanghai Ocean University, 24, 203-210. (in Chinese with English abstract) |
| [席晓晴, 鲍宝龙, 章守宇 (2015) DNA条形码在鱼类胃含物种类鉴定中的应用. 上海海洋大学学报, 24, 203-210.] | |
| [29] | Yang MJ, Feng J, Yu ZL, Song H, Hu Z, Zhou C, Shi P, Zhang T (2021) Comparative analysis of the feeding habits of Rapana venosa and Neptunea arthritica cumingii near Zhangzi Island, China, based on stable isotope ratios and fatty acid profiles. Aquaculture Research, 52, 1846-1854. |
| [30] | Zhang CC, Zhang DD, Wang ZQ, Luo FG, Wen YH, Jiang M, Li YH (2023) Microscope combined with high-throughput sequencing to analyze the stomach content of Bellamya aeruginosa. Freshwater Fisheries, 53(2), 84-93. (in Chinese with English abstract) |
| [张灿灿, 张丹丹, 王志强, 罗福广, 文衍红, 蒋明, 李艳和 (2023) 基于镜检和高通量测序技术的铜锈环棱螺胃含物分析. 淡水渔业, 53(2), 84-93.] | |
| [31] | Zhang DD, Dong XY, Zhu JY, Yang JC, Tian Y, Wang L, Mao J, Wang XB, Chang YQ, Hao Z (2021) Effect of water temperature on the behavior of Neptunea cumingii and the histology, immune enzyme activity, and transcriptome of its gills and kidneys. Invertebrate Survival Journal, 19, 1-12. |
| [32] | Zhang HY, Xu Q, Zhao Y, Yang HS (2016) Sea cucumber (Apostichopus japonicus) eukaryotic food source composition determined by 18s rDNA barcoding. Marine Biology, 163, 153. |
| [33] | Zhang YY, Dong JY, Sun X, Zhang XM (2022) A review of bibliometric analysis of feeding habits on DNA-based molecular dietary research. Fisheries Science, 41, 160-172. (in Chinese with English abstract) |
| [张宇洋, 董建宇, 孙昕, 张秀梅 (2022) 基于DNA分子生物学食性研究领域的文献计量分析. 水产科学, 41, 160-172.] | |
| [34] | Zhou TC, Hu SM, Lin XZ, Liu S, Huang H (2020) Study on the feeding habits of Tectus pyramis in the coral reef ecosystem based on 18S rDNA barcoding. Marine Sciences, 44(2), 99-107. (in Chinese with English abstract) |
| [周天成, 胡思敏, 林先智, 刘胜, 黄晖 (2020) 基于18S rDNA条形码技术的珊瑚礁区塔形马蹄螺(Tectus pyramis)食性分析. 海洋科学, 44(2), 99-107.] |
| [1] | 董志远, 陈琳琳, 张乃鹏, 陈莉, 孙德斌, 倪艳梅, 李宝泉. 基于环境DNA宏条形码技术研究黄河三角洲典型潮沟系统鱼类多样性及其对水文连通性的响应[J]. 生物多样性, 2023, 31(7): 23073-. |
| [2] | 湛振杰, 张超, 陈敏豪, 王嘉栋, 富爱华, 范雨薇, 栾晓峰. 基于DNA宏条形码技术的大兴安岭北部欧亚水獭冬季食性分析[J]. 生物多样性, 2023, 31(6): 22586-. |
| [3] | 彭步青, 陶玲, 李靖, 范荣辉, 陈顺德, 付长坤, 王琼, 唐刻意. 基于DNA宏条形码研究四川老君山国家级自然保护区6种同域共存小型哺乳动物的食性[J]. 生物多样性, 2023, 31(4): 22474-. |
| [4] | 沈梅, 郭宁宁, 罗遵兰, 郭晓晨, 孙光, 肖能文. 基于eDNA metabarcoding探究北京市主要河流鱼类分布及影响因素[J]. 生物多样性, 2022, 30(7): 22240-. |
| [5] | 陆琪,胡强,施小刚,金森龙,李晟,姚蒙. 基于分子宏条形码分析四川卧龙国家级自然保护区雪豹的食性[J]. 生物多样性, 2019, 27(9): 960-969. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
备案号:京ICP备16067583号-7
Copyright © 2022 版权所有 《生物多样性》编辑部
地址: 北京香山南辛村20号, 邮编:100093
电话: 010-62836137, 62836665 E-mail: biodiversity@ibcas.ac.cn
![]()