生物多样性是维持生态系统稳定性的基础, 也是影响生态系统生产力、抗干扰能力及恢复能力等功能的重要因素(Hooper et al, 2005; Cardinale et al, 2012)。在衡量生物多样性的三个层次即遗传多样性、物种多样性和生态系统多样性中, 物种多样性是对一个自然地理分布区域生物多样性情况最直观的反映, 物种的组成和结构对种群动态及生态系统功能有重要决定作用。在一个生态系统中共存的各物种通过直接或间接的取食与被取食关系交织形成食物网, 不同物种间的复杂相互作用促进生态系统功能的正常运转, 进而维持着生态系统的稳定性和物种的多样性(Montoya et al, 2003)。对世界不同地区和多种生态系统类型的研究普遍显示, 物种多样性的丧失是生态环境退化和生态系统失衡的主要因素(Hooper et al, 2012)。因此, 对物种多样性及其相互作用的了解是认识生态系统过程的重要前提, 也是保护生态环境和生物多样性的基础。
大型哺乳类食肉动物(以下简称大型食肉动物)处于陆地生态系统食物链的顶端, 通过捕食、竞争、威慑和食物网级联效应调控大型食草动物、中型食肉动物及其他食物网成员的种群数量和动态, 深刻影响食物网结构及生态系统功能(Wang & Macdonald, 2009; Wang et al, 2014)。多项研究均显示, 大型食肉动物从原来的生物群落中消失后会引发一系列下层营养级动物种群乃至植被和无机环境的重大变化, 对生态系统产生不可逆转的影响(Ripple et al, 2014)。在世界范围内, 由于一个多世纪以来人口的迅速增长和生产生活范围扩大, 大型食肉动物原生栖息地丧失和破碎化, 再加上捕猎以及细菌、病毒等病原体的传播, 目前其种群生存普遍面临严重的威胁, 多种食肉动物数量大幅度减少, 分布的地理范围急剧收缩(Ripple et al, 2014)。如何有效保护大型食肉动物物种多样性及种群数量已经成为世界关注的焦点问题和保护生物学的重要研究方向。
我国西南山地北部与青藏高原东缘交界地带的川西高原属世界生物多样性热点地区之一(Myers et al, 2000), 因其特殊的自然历史、地理条件及气候特征, 为多种野生动物提供了适宜的栖息地环境, 其哺乳类和鸟类数量分别占全国的20%和33%, 有12种兽类和10种鸟类被列为国家一级重点保护野生动物, 23种兽类和49种鸟类被列为国家二级重点保护野生动物。该区域也是世界范围内大型食肉动物物种最为丰富的地区之一(Ripple et al, 2014)。川西高原独特的自然地理环境和丰富的生物多样性使其具有极高的生态学研究和保护价值, 而目前对该地区物种分布情况及生态系统过程了解甚少。获得准确的物种多样性信息及食肉动物食性数据有助于深入了解该地区生态系统结构及食物网关系, 对研究物种共存机制及生物多样性保护有重要意义。与低海拔的热带和亚热带森林生态系统相比, 由于气候条件恶劣、水热资源缺乏、生物量和生产力低, 高原生态系统更为脆弱(Cole & Landres, 1996); 加之该地区人类活动不断加强, 有可能加剧对当地动植物资源的破坏(Baker et al, 2008), 因此对这一生态系统的研究和保护迫在眉睫。
物种和种群调查是有效开展濒危物种保护与管理工作的前提和基础(Bellemain et al, 2010)。由于食肉目动物活动范围广, 多夜间行动且警觉性高, 在野外很难对其进行直接观测, 使对此类动物的调查和保护工作更加困难(Pelton et al, 2003)。传统的食肉动物物种识别方法主要有足迹识别法、毛发形态识别法、咬痕识别法等依据形态学和解剖学特征的鉴定方法(周用武和杨玉华, 2009; 张帅等, 2013)。由于近缘物种形态相似度高, 而同种动物受生境条件、发育阶段及性别等因素的影响可能存在个体形态差异, 此外还容易受到研究者经验等主观因素的干扰, 因此形态学鉴定方法通常存在定种困难、错误率高、效率低、结果难以验证等问题(程希婷等, 2011)。
动物的捕食行为是重要的生态过程, 对食性的分析及食物网络的研究可以直接体现出生态群落的功能和结构, 揭示各级消费者之间的能量流动和物质利用转换, 对捕食者在群落中的作用有更深理解, 是生态系统研究的重要组成部分(王雪芹等, 2015), 对动物食性的了解还可以为保护生态系统功能及物种多样性提供科学依据。对野生动物食性的研究方法传统上主要依赖于形态学分析, 包括直接观察法、胃内容物分析法、粪便显微镜观察法等(尹华宝等, 2008)。但这些方法受限于食物类型和消化情况, 并且可能存在多种形态相似难以区分的近缘种, 需要分析人员有丰富的生物学分类经验, 鉴定结果的效率和准确性较低(Pompanon et al, 2012)。
近几十年来分子生物学和生物信息学技术的飞速发展, 推动DNA分析成为生态学和保护生物学研究的重要工具。尤其是DNA条形码(DNA barcoding)技术(即利用具有种内特异性和种间变异性的相对较短的标准DNA片段, 建立一种新的物种身份识别系统), 可以对物种进行快速、准确的识别和鉴定, 成为物种多样性调查的有力手段(Hebert et al, 2003; Valentini et al, 2009; Kress et al, 2015)。二代测序技术的不断进步使得高通量、低成本地获取大量样品的DNA序列信息成为可能。DNA宏条形码(metabarcoding)技术就是利用高通量测序技术大量获得混合样本中多种条形码基因扩增子序列, 继而通过生物信息学手段分析实现分类单元鉴定的方法(Creer et al, 2010; Porazinska et al, 2010; Voolstra et al, 2011)。DNA宏条形码以高通量、低成本、快速鉴定大量物种的优势, 极大地拓展了DNA条形码应用的范围(Taberlet et al, 2012; 马兰, 2013)。在生态学领域, DNA宏条形码已广泛应用于生物多样性分析、食性分析、古生物研究、生态评估等多方面(Pompanon et al, 2012; Yoccoz et al, 2012; Murray et al, 2013; Kelly et al, 2014)。基于DNA宏条形码的分子食性分析方法通过从样品(如粪便)中提取DNA, 设计针对某一种、某几种物种或某一类群的引物, 利用聚合酶链式反应(PCR)扩增特定的条形码序列, 并通过高通量测序和序列比对来识别食物种类(Valentini et al, 2009)。与基于显微观察的食性分析方法相比, 这种方法的优势在于: (1)不依赖于研究者的形态学经验和主观判断; (2)对形态相似的近缘种和隐存种可以准确区分; (3)不受食物消化程度和硬质残留的局限, 可以分析几乎所有食物类型(如肌肉、内脏等); (4)具有极高的灵敏度, 微量的食物DNA也可以被检测到; (5)能对混合DNA样品进行测序且通量高, 大大降低了分析成本和时间(Symondson, 2002; Valentini et al, 2009; Kircher & Kelso, 2010)。
本研究拟通过基于野外采集食肉动物粪便DNA的物种鉴定, 获得位于我国西南山地的四川甘孜藏族自治州的食肉动物物种多样性初步信息, 并基于高通量测序和DNA宏条形码技术, 进行食肉动物精准食性分析。这些信息有助于深入了解研究地区生态系统食物网结构及功能, 对研究物种共存机制及生物多样性保护有重要意义。同时本研究使用的粪便DNA及宏条形码技术为脊椎动物多样性快速调查及高通量精确食性分析提供了有力借鉴。
1 研究方法
1.1 研究区域
甘孜藏族自治州(以下简称甘孜州)位于青藏高原东南缘四川省西南部, 介于97°22°-102°29° E、27°58°-34°20° N之间, 面积153,000 km2, 辖18个县。全州地貌大致分为: 山地、丘状高原、山原、高山峡谷等4种类型, 平均海拔3,500 m以上。该地区纬度位置属于亚热带气候区, 由于受到青藏高原复杂地形的影响, 境内气候呈现出青藏高原山地气候和大陆性气候特征, 属大陆性季风高原型气候。全州年平均气温8-10℃, 日照多、辐射强, 昼夜温差大, 干湿季明显, 年降水量300-600 mm, 水热同期。甘孜州总林地面积17,200 km2, 林草覆盖率达83%。
1.2 样品采集与保存
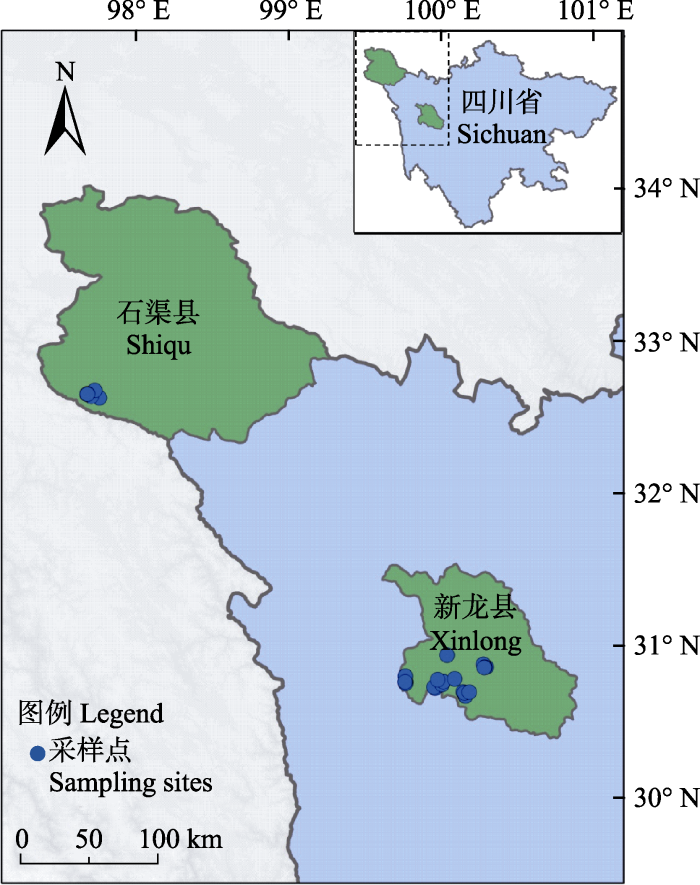
本研究粪便样品于2016年9-10月采集自甘孜州新龙县及石渠县境内(图1)。由调查人员沿野外样线进行搜索, 发现新鲜食肉动物粪便时, 首先用GPS手持机(Garmin 60CXS)记录采集时间及地理信息, 然后用一次性PE手套拾取单个粪便样品装入50 mL的无菌样品管中, 并在管盖和管身记录样品编号、详细地名及根据粪便形态鉴定的物种。同时填写“野外动物样品记录卡”, 包括编号、样品类型、生境类型等详细信息。在每次拾取样品时更换新的手套以避免交叉污染。当日将采集的样品浸没在无水乙醇中, 24 h后将乙醇倒掉, 倒入硅胶干燥保存。
图1
图1
四川省甘孜藏族自治州石渠县及新龙县采样地点示意图
Fig. 1
Map of sampling area in Shiqu and Xinlong counties of Ganzi Tibetan Autonomous Prefecture, Sichuan
1.3 粪便DNA提取、扩增及物种鉴定
本研究使用适用于食肉动物粪便的2CTAB- PCI法(Vallet et al, 2007)提取和纯化DNA, 并根据食肉动物粪便特点进行了若干改进(Xiong et al, 2017), 取样时从粪便内部多点夹取, 粪便总量不超过500 μL, 并捣碎均匀, 使其能充分反应, 提取过程中用CTAB及淀粉去除DNA中的PCR抑制物, 并用苯酚氯仿多次抽提DNA以达到提纯DNA的目的。
首先对每份样品的来源物种(即捕食者)进行分子物种鉴定。由于在粪便DNA中食物DNA降解较严重, 难以扩增长片段, 而捕食者自身DNA相对质量较好, 故首先使用脊椎动物通用引物16S-F/R (Xiong et al, 2016)(附录1)扩增长380 bp左右的捕食者线粒体16S片段。每个样品PCR总体系为30 μL, 包含DNA (10-100 ng/μL) 6 μL、2 × EasyTaq PCR SuperMix (全式金, AS111) 15 μL、正反向引物各0.2 μM及0.4 mg/mL牛血清白蛋白(BSA)。PCR反应程序为95℃预变性5 min; 然后95℃变性30 s, 55℃退火30 s, 72℃延伸30 s, 进行35个循环; 最后72℃延伸10 min。纯化后的PCR产物使用ABI PRISM3730xl测序仪(Applied Biosystems, Foster City, California)进行测序。测序结果通过软件Chromas v2.22 (Technelysium Pry Ltd., Australia)处理, 得到长度约320 bp的单一序列。然后用NCBI (National Center for Biotechnology Information,
1.4 高通量测序及数据处理
确定粪便来源物种后, 进一步针对不同食肉动物进行基于DNA宏条形码的分子食性分析。粪便样品中残存的被捕食者DNA较捕食者自身DNA量少且破碎化严重, 所以为了保证被捕食者DNA的成功扩增, 食性分析通用引物的扩增产物长度不宜超过150 bp。选用Riaz等(2011)针对脊椎动物设计的12SV5-F/R通用引物(附录1), 并在正反向引物5°端增加7个核苷酸的引物标签(Coissac, 2012)进行PCR扩增, 产物长度约140 bp。每个PCR采用具有不同标签序列的引物扩增, 后续高通量测序结果根据标签进行样品区分。该引物用ecoPCR软件通过数据库模拟PCR及多种食肉动物粪便样品进行验证, 通用性广且具有较高的脊椎动物物种分辨率, 对啮齿目、兔形目和鸡形目的物种鉴定表现佳(Shehzad et al, 2012a, b; Xiong et al, 2017)。粪便样品总DNA中同时包括捕食者与被捕食者DNA, 使用脊椎动物通用引物能够同时扩增二者的序列, 由于捕食者自身DNA占较多量, 扩增时会与被捕食者DNA竞争引物而影响食物DNA的扩增效率及成功率(Pompanon et al, 2012)。本文使用文献报道的豹猫(Prionailurus bengalensis)序列特异性阻抑引物(Shehzad et al, 2012a), 并自行设计了其他食肉动物物种的阻抑引物(附录1), 通过将各阻抑引物与当地主要物种的相应区段进行比较, 可能的被捕食者均与这段阻抑引物序列具有多于10个核苷酸位点的差异。阻抑引物浓度过高会影响PCR产物中被捕食者序列的扩增(Vestheim & Jarman, 2008)。经过前期预实验, 最终确定在反应体系中阻抑引物与通用引物的工作浓度比为10 : 1。对28份鉴种成功的食肉动物粪便每个样品的DNA进行3次PCR平行对照反应, 每个反应分别使用不同标签的通用引物加以区分。每个样品PCR总体系为30 μL, 包含DNA (10-100 ng/μL) 6 μL、2 × EasyTaq PCR SuperMix 15 μL、正反向引物各0.2 μM、阻抑引物2 μM及0.4 mg/mL BSA。PCR反应程序为95℃预变性5 min; 然后95℃变性30 s, 60℃退火30 s, 进行35个循环; 最后72℃延伸10 min。为检验本底污染和交叉污染情况, 每批次扩增中包括DNA提取空白对照和不加模板的PCR空白对照反应, 共3个DNA提取空白对照和21个PCR空白对照。全部样品及对照反应共计108个PCR反应。
将所有PCR产物进行纯化并混合, 在北京诺和致源生物科技有限公司进行高通量测序。测序采用Paired-end法, 使用Illumina HiSeq2500测序平台(Illumina Inc., San Diego, CA, USA), 单端读段长度为150 bp。高通量数据的处理参考网站
当某一序列在本底数据库中最佳匹配序列多于1条时, OBITools中的程序会自动将物种鉴定结果记为能够涵盖所有匹配物种的最低分类单元, 但不能加入研究地物种分布信息的参考。结果中还可能存在污染及PCR错误, 或者未能确定物种的序列, 所以需要在OBITools输出结果的基础上进行人工排查, 必要时用BLAST程序检索目标片段的匹配物种。
序列的物种鉴定原则如下:
(1)与单一物种比对的一致度 ≥ 99%, 且符合物种分布记录, 记为该物种; 如发生与单一物种比对一致度 ≥ 99%但该物种非当地物种时, 则记为该物种在当地有分布的最近缘物种。
(2)与多个物种比对的一致度 ≥ 99%时, 首先排除当地无分布的物种, 如果仍不能确定为单一物种, 则记为能够涵盖所有一致度 ≥ 99%的当地物种的最低分类单元。
(3)比对结果中, 最高一致度 < 99%且 ≥ 95%时, 一致度最高序列仅有1条, 且与得分次之的序列一致度差距 ≥ 2%时, 记为一致度最高序列的上一级分类单元。最高一致度对应有多个物种时, 则记为能够涵盖所有一致度最高的当地物种的最低分类单元。
(4)如果最高一致度 ≥ 95%范围内仅对应1种非当地物种, 则记为当地有记录的该物种最近缘种。
(5)比对结果中, 最高一致度 < 95%时, 认为无法判定该序列的分类单元, 记为“未知”。
依据上述原则对每一序列进行物种鉴定与分类, 物种分布信息参考《中国兽类野外手册》(Smith和解焱, 2009)。由于条形码分辨率及数据库的局限, 不是每条序列的识别都能达到物种水平, 所以我们使用分子可操作分类单元(molecular operational taxonomic unit, MOTU)的概念来对不同食物类群进行区分(Pompanon et al, 2012)。
1.5 食性分析
首先, 对食物MOTU进行筛选与整理。当单一PCR产物中某食物MOTU序列数少于同批PCR空白对照或DNA提取空白对照, 或单一PCR产物中某食物MOTU序列数少于该PCR总序列数的1%时, 认为该序列可能来源于交叉污染, 该MOTU不计入相应样品的食物结果。在每个样品中保留至少在2个PCR平行对照中出现的MOTU。序列处理中舍弃与GenBank比对后最高一致度低于95%的序列, 并合并彼此相差小于2% (即相差1个核苷酸)的序列。物种鉴定时出现的食肉动物序列及可能来自实验污染的人的序列不计入食物MOTU。经过以上筛选和处理后, 在每个样品中每一MOTU出现赋值为1, 反之记为0, 统计各食物MOTU的出现频次。
使用%FO和%RO两个指标估计各食肉动物的食性构成(Xiong et al, 2017)。对某一食肉物种而言, %FO是指含某食物MOTU的样品数占总样品数的百分比, 计算公式如下:
其中, Ni表示出现了第i类食物MOTU的样品数(次数), N是有效样品总数。
%RO是某食物MOTU的出现频次占所有食物MOTU出现总频次的百分比, 计算公式如下:
其中, Ni含义同上, $\Sigma_{N_i}$表示该物种所有食物MOTU出现次数之和。
单一物种全部粪便样品中所有食物MOTU的%RO之和为1, 而当存在1个或多个样品中检出不止1种食物MOTU时, 该物种的%FO之和大于1。%FO表示某个食物MOTU在该物种食性中出现的常见程度, %RO则侧重显示该食物MOTU在食性中所占份额和重要性(Wang et al, 2014; Gómez-Ortiz et al, 2015; Kasper et al, 2016)。
2 结果
2.1 样品采集及物种鉴定
于2016年秋季在石渠县(6份)和新龙县(32份)共采集38份食肉动物粪便样品, 分别进行DNA提取, 其中28份可以被通用引物16S-F/R扩增并成功判断出样品来源物种, 物种鉴定成功率为73.7%。其余10份样品未能成功获得扩增产物进行测序及物种鉴定。
共鉴定出7种食肉动物(表1), 包括狼(Canis lupus, N = 8)、狗(C. lupus familiaris, N = 6)、豹猫(N = 6)、赤狐(Vulpes vulpes, N = 3)、棕熊(Ursusarctos, N = 3)、豹(Panthera pardus, N = 1)和雪豹(P. uncia, N = 1)。
28份物种鉴定成功的样品中, 有22份在样品采集时由野外工作人员根据粪便形态进行了物种预判, 其中仅有8份样品预判物种与分子鉴定结果相同, 且各食肉动物粪便的预判成功率各不相同, 总体预判正确率仅为36.4% (表1)。
表1 物种分子鉴定结果及形态物种预判正确率
Table 1
| 物种 Species | 样品数 No. of sample | 物种预判 样品数 No. of morphologically identified sample | 预判正确 样品数 No. of correctly identified sample | 预判正确率 Accuracy of morphological identification (%) | |
|---|---|---|---|---|---|
| 狼 Canis lupus | 8 | 4 | 1 | 25.0 | |
| 狗 Canis lupus familiaris | 6 | 6 | 1 | 16.7 | |
| 豹猫 Prionailurus bengalensis | 6 | 4 | 4 | 100 | |
| 赤狐 Vulpes vulpes | 3 | 3 | 0 | 0 | |
| 棕熊 Ursus arctos | 3 | 3 | 0 | 0 | |
| 豹 Panthera pardus | 1 | 1 | 1 | 100 | |
| 雪豹 Panthera uncia | 1 | 1 | 1 | 100 | |
| 合计 Total | 28 | 22 | 8 | 36.4 | |
2.2 食性分析
高通量测序结果经过OBITools软件的过滤和降噪, 并对这些序列进一步进行人工筛选和处理。序列分析结果显示所有28份样品均得到有效食性数据, 共计28个不同的食物MOTU。其中17个MOTU鉴定到物种水平, 占MOTU总数的60.7%, 其他MOTU分别被鉴别至属、科及目水平(表2)。
表2 食物分子可操作分类单元(MOTU)中不同分类级别的数目及比例
Table 2
| 分类等级 Taxonomic level | 种 Species | 属 Genus | 科 Family | 目 Order | 总计Total |
|---|---|---|---|---|---|
| MOTU数目 MOTU no. | 17 | 8 | 2 | 1 | 28 |
| 比例 Percentage (%) | 60.7 | 28.6 | 7.1 | 3.6 |
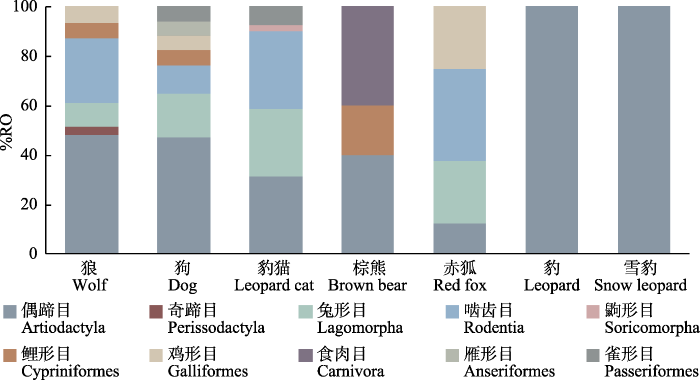
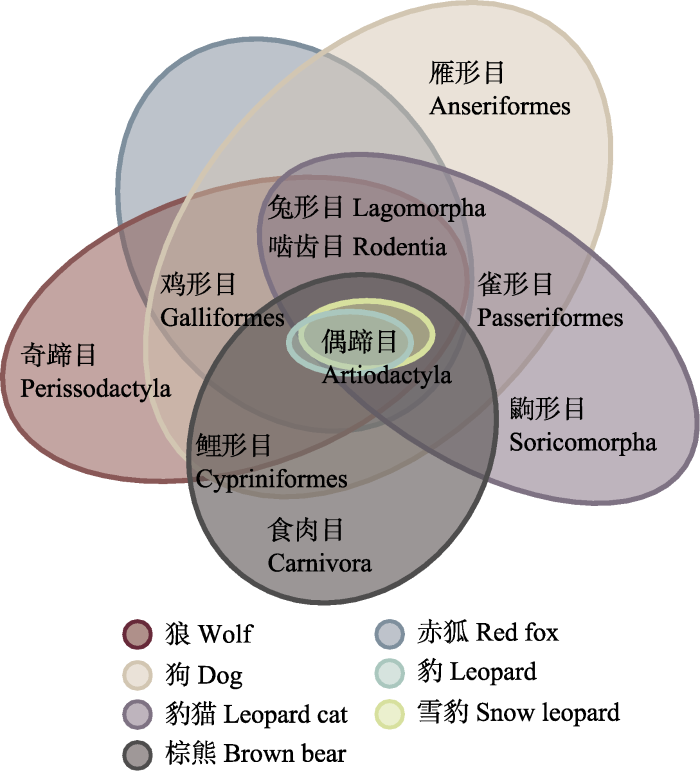
28个食物MOTU包括19种哺乳类、8种鸟类和1种鱼类, 分属于10个目(表3)。大型食肉动物狼、狗和棕熊的食物中偶蹄目均占最大比例, %RO分别为48%、47%和40% (图2); 而狼和狗又体现了更多的食物多样性和相似性, 家牦牛(Bos grunniens)是它们食物中出现频率最高的物种, %FO分别为75%和67%, 除偶蹄目之外, 它们都取食啮齿目、兔形目、鲤形目和鸡形目动物(图2, 3)。除偶蹄目外, 棕熊较多以食肉目的猪獾(Arctonyx albogularis)作为食物(表3)。中小型食肉动物豹猫和赤狐的食物中小型兽类如啮齿目和兔形目占主要比例, 其中出现频率最高的是啮齿目, 在豹猫和赤狐食性构成中%RO分别为34%和38%, 兔形目其次, %RO分别为27%和25% (图2)。其中高原松田鼠(Neodon irene)和高原鼠兔(Ochotona curzoniae)是豹猫和赤狐主要取食的啮齿类和兔形目食物(表3)。与赤狐相比, 豹猫的食物多样性更高, 它还会较多取食如高原兔(Lepus oiostolus)、姬鼠属(Apodemus)、白腹鼠属(Niviventer)、鼯鼠属(Petaurista)等, 同时鼩鼱属(Sorex)只在豹猫食物中出现(图3); 赤狐除捕食小型哺乳类外还取食鸡形目动物, 如白马鸡(Crossoptilon crossoptilon)和山鹑属(Perdix)。与狼和狗相比, 豹猫和赤狐食物中各目动物的出现比例更为平均。豹和雪豹的样品仅各1份, 各只鉴定出1种食物, 豹的食物为中华斑羚(Naemorhedus griseus), 雪豹的食物为岩羊(Pseudois nayaur)。食肉动物物种间食物重叠情况见图3。
表3 7种食肉动物食物分子可操作分类单元(MOTU)分子鉴定结果。最佳匹配物种结果中斜线(/)用于分隔序列一致度相同的物种, 竖线(|)用于分隔一致度相差 ≥ 1%的物种。
Table 3
| 食物 MOTU Food MOTU | 在不同食肉动物样品中的出现频次 Occurrence frequency in different carnivore samples | GenBank 最佳匹配物种 Best match species in GenBank | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 拉丁学名 Scientific name | MOTU 名称 MOTU name | 狼 Wolf | 狗 Dog | 豹猫 Leopard cat | 棕熊 Brown bear | 赤狐 Red fox | 豹 Leopard | 雪豹 Snow leopard | 中文名 Chinese name | 拉丁名 Scientific name | 最高一致度 Best identity (%) | 序列号 Accession no. | ||||||||||||||
| N = 8 | N = 6 | N = 6 | N = 3 | N = 3 | N = 1 | N = 1 | ||||||||||||||||||||
| 偶蹄目 Artiodactyla | ||||||||||||||||||||||||||
| Bos grunniens | 家牦牛 | 6 | 4 | 6 | 1 | 0 | 0 | 0 | 家牦牛 | Bos grunniens | 100 | KX232527.1 | ||||||||||||||
| Elaphodus cephalophus | 毛冠鹿 | 2 | 2 | 3 | 0 | 0 | 0 | 0 | 毛冠鹿 | Elaphodus cephalophus | 99 | DQ873526.1 | ||||||||||||||
| Sus scrofa | 野猪 | 2 | 2 | 3 | 0 | 0 | 0 | 0 | 野猪 | Sus scrofa | 100 | KX886757.1 | ||||||||||||||
| Pseudois nayaur | 岩羊 | 2 | 0 | 0 | 0 | 1 | 0 | 1 | 岩羊 | Pseudois nayaur | 100 | KP998469.1 | ||||||||||||||
| Moschus | 麝属 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 马麝/林麝 | Moschus chrysogaster/ Moschus berezovskii | 100 | KP684123.1/ AY184425.1 | ||||||||||||||
| Przewalskium albirostris | 白唇鹿 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 白唇鹿 | Przewalskium albirostris | 100 | JN632690.1 | ||||||||||||||
| Naemorhedus griseus | 中华斑羚 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 中华斑羚 | Naemorhedus griseus | 100 | JN632664.1 | ||||||||||||||
| 奇蹄目 Perissodactyla | ||||||||||||||||||||||||||
| Equus caballus | 马 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 马 | Equus caballus | 100 | KU575247.1 | ||||||||||||||
| 兔形目 Lagomorpha | ||||||||||||||||||||||||||
| Ochotona curzoniae | 高原鼠兔 | 2 | 3 | 5 | 0 | 2 | 0 | 0 | 高原鼠兔 | Ochotona curzoniae | 100 | KM225732.1 | ||||||||||||||
| Lepus oiostolus | 高原兔 | 1 | 0 | 6 | 0 | 0 | 0 | 0 | 高原兔 | Lepus oiostolus | 99 | AY745187.1 | ||||||||||||||
| 啮齿目 Rodentia | ||||||||||||||||||||||||||
| Neodon irene | 高原松田鼠 | 4 | 1 | 5 | 0 | 2 | 0 | 0 | 高原松田鼠 | Neodon irene | 100 | HQ416908.1 | ||||||||||||||
| Apodemus | 姬鼠属 | 2 | 0 | 5 | 0 | 0 | 0 | 0 | 大耳姬鼠| 高山姬鼠 | Apodemus latronum| Apodemus chevrier | 100| 99 | HQ333256.1| HQ896683.1 | ||||||||||||||
| Marmota himalayana | 喜马拉雅 旱獭 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 喜马拉雅 旱獭 | Marmota himalayana | 100 | JX069958.1 | ||||||||||||||
| Niviventer excelsior | 川西白腹鼠 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 川西白腹鼠 | Niviventer excelsior | 99 | JQ927552.1 | ||||||||||||||
| Trogopterus xanthipes | 复齿鼯鼠 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 复齿鼯鼠 | Trogopterus xanthipes | 97 | AY227546.1 | ||||||||||||||
| Petaurista | 鼯鼠属 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 红背鼯鼠/ 霜背大鼯鼠| 红白鼯鼠 | Petaurista petaurista/ Petaurista philippensis| Petaurista alborufus | 98| 97 | KP973556.1/ KP973555.1| AY227541.1 | ||||||||||||||
| Eozapus setchuanus | 林跳鼠 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 林跳鼠 | Eozapus setchuanus | 98 | KJ648495.1 | ||||||||||||||
| 食物MOTU Food MOTU | 在不同食肉动物样品中的出现频次 Occurrence frequency in different carnivore samples | GenBank最佳匹配物种 Best match species in GenBank | ||||||||||||||||||||||||
| 拉丁学名 Scientific name | MOTU 名称 MOTU name | 狼 Wolf | 狗 Dog | 豹猫 Leopard cat | 棕熊 Brown bear | 赤狐 Red fox | 豹 Leopard | 雪豹 Snow leopard | 中文名 Chinese name | 拉丁名 Scientific name | 最高一致度 Best identity (%) | 序列号 Accession no. | ||||||||||||||
| N = 8 | N = 6 | N = 6 | N = 3 | N = 3 | N = 1 | N = 1 | ||||||||||||||||||||
| 食肉目 Carnivora | ||||||||||||||||||||||||||
| Arctonyx albogularis | 猪獾 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 猪獾 | Arctonyx albogularis | 100 | HM106329.1 | ||||||||||||||
| 鼩形目 Soricomorpha | ||||||||||||||||||||||||||
| Sorex | 鼩鼱属 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 纹背鼩鼱/ 小纹背鼩鼱| 长爪鼩鼱/ 普通鼩鼱 | Sorex cylindricauda/ Sorex bedfordiae| Sorex unguiculatus/ Sorex araneus | 99| 98 | KF696672.1/ GU981054.1| KX754508.1/ KT210896.1 | ||||||||||||||
| 鲤形目 Cypriniformes | ||||||||||||||||||||||||||
| Cyprinidae | 鲤科 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 汪氏近红鲌/ 厚颌鲂 | Ancherythroculter wangi/ Megalobrama pellegrini | 100 | MG783573.1/ JX242529.1 | ||||||||||||||
| 鸡形目 Galliformes | ||||||||||||||||||||||||||
| Crossoptilon crossoptilon | 白马鸡 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | 白马鸡 | Crossoptilon crossoptilon | 100 | KP259808.1 | ||||||||||||||
| Gallus gallus | 家鸡 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 原鸡 | Gallus gallus | 100 | KX987152.1 | ||||||||||||||
| Perdix | 山鹑属 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 斑翅山鹑/ 高原山鹑/ 灰山鹑 | Perdix dauurica/ Perdix hodgsoniae/ Perdix perdix | 100 | KY411596.1/ KF027440.1/ KF781322.1 | ||||||||||||||
| 雁形目 Anseriformes | ||||||||||||||||||||||||||
| Anas | 鸭属 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 绿头鸭/ 琵嘴鸭/ 绿翅鸭/ 斑嘴鸭/ 针尾鸭 | Anas platyrhynchos/ Anas clypeata/ Anas crecca/ Anas poecilorhyncha/ Anas acuta | 100 | KX592536.1/ KT345702.1/ KC771255.1/ KC466567.1/ KF312717.1 | ||||||||||||||
| 雀形目 Passeriformes | ||||||||||||||||||||||||||
| Phylloscopus 1 | 柳莺属1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 暗绿柳莺| 淡眉柳莺 | Phylloscopus trochiloides| Phylloscopus humei | 99| 98 | KP267717.1| KP267716.1 | ||||||||||||||
| Phylloscopus 2 | 柳莺属2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 褐柳莺/ 黄腰柳莺/ 乌嘴柳莺 | Phylloscopus fuscatus/ Phylloscopus proregulus/ Phylloscopus maculipennis | 100 | JF505332.1/ AY635103.1 /AY635100.1 | ||||||||||||||
| Muscicapidae 1 | 鸫科 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 鹊鸲/ 北红尾鸲 | Copsychus saularis/ Phoenicurus auroreus | 99 | KU058637.1/ KF997864.1/ KF997863.1 | ||||||||||||||
| Passeriformes | 雀形目 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 家燕/ 红喉歌鸲 | Hirundo rustica/ Luscinia calliope | 96 | KX398931.1/ HQ690246.1 | ||||||||||||||
此外, 家畜(家牦牛、马Equus caballus)在4种食肉动物中出现, 且狼、狗和棕熊取食家畜的%RO都在20%以上。
图2
图2
目水平上不同食肉动物食物组成。%RO为不同食物分子可操作分类单元(MOTU)出现的相对频率。
Fig. 2
Dietary compositions by prey order identified in the diets of different carnivores. %RO indicates the relative frequency of occurrence of different food MOTUs.
图3
3 讨论
3.1 食肉动物物种鉴定
本研究在甘孜藏族自治州新龙县和石渠县通过野外粪便DNA进行物种鉴定, 共发现食肉动物7种, 分别为狼、棕熊、豹、雪豹、豹猫、赤狐等6种野生食肉动物及1种家养食肉动物狗, 为这些物种在当地的分布提供了确凿的证据, 同时显示了基于粪便DNA进行物种快速调查的高效和便捷性。
形态学物种鉴定方法可通过粪便的外形、颜色、气味、内容物等特征来判断来源物种, 此类方法受到多种物种自身因素、环境条件以及样品采集者的经验和主观判断的影响, 通常存在较高的错误率(Monterroso et al, 2013; Lonsinger et al, 2015)。本次样品采集时, 有22份样品由野外调查人员进行了物种预判, 但仅有8份与分子鉴定结果一致, 即传统物种鉴定方法的正确率为36.4% (表1)。由此可见, 基于DNA条形码的分子鉴定方法大大提高了由粪便进行物种识别的准确率。但就本次物种鉴定结果来看, 可能由于粪便样品质量或者PCR抑制物过多, 38份粪便样品中, 只有28份可以成功提取DNA并进行PCR扩增测序, 物种识别成功率为73.7%。这一问题可以通过优化DNA提取方法和后续采集样品时尽量采集新鲜粪便保证样品质量来提高成功率。
3.2 食肉动物的食性组成
用DNA宏条形码和高通量测序相结合的方法, 从28份粪便样品中获得了28个食物MOTU, 快速展示了该地区7种食肉动物的部分食物物种组成。由于赤狐、棕熊、豹和雪豹的样品数均较少(≤ 3), 所以仅对样品数量相对较多的狼、狗和豹猫进行讨论。
北美及欧洲的大量研究表明, 有蹄类是狼的主要食物, 这与能量收益率和猎物的可利用度有关(Marfuard, 1998)。目前我们的结果显示, 狼取食最多的是偶蹄目动物, 其中家牦牛在食物组成中出现频率最高, 说明狼可能是对家畜危害最严重的食肉动物之一。原因可能是当地家牦牛数量远大于野生有蹄类, 使其成为狼的主要捕食目标, 但不排除部分来源是食腐(取食死亡家畜残骸)或捡拾人类丢弃食物残余的可能。这一结果与我国其他很多地区相似, 比如在内蒙古地区狼食性研究中, 家畜在狼食谱中的频率(%RO)高达60%以上(颜文博等, 2006)。在青海, 狼食性中家畜的比例(%RO)从38%到78%不等(Liu & Jiang, 2003)。据国内外狼食性研究, 狼在枯草期还会捕食啮齿类和兔类作为补充(张洪海等, 2000; 颜文博等, 2006), 是被捕食比例仅次于有蹄类的猎物类型, 在我们的结果中啮齿目和兔形目物种也占狼食物的较大比例。而啮齿目比兔形目出现频率高, 原因可能是在研究地区啮齿目动物数量更多且易捕食, 或是啮齿目动物生物量远小于兔形目动物, 需捕食更多个体才能满足营养需求。
甘孜州以农牧业生产方式为主, 藏民多养狗来看家和放牧。由于宗教信仰, 藏民不杀生, 对伤人的狗和数量过剩的狗会进行驱逐而不是将其杀死, 因此许多具有獒血统的逃逸和散养家狗汇集成群, 穿梭于人类生活区与野生动物栖息地, 很可能会与野生食肉动物直接竞争生存资源(Mitchell & Banks, 2005; Glen et al, 2006; Glen & Dickman, 2008)。本研究结果中, 狗的食物中包括毛冠鹿(Elaphodus cephalophus)、野猪(Sus scrofa)、高原鼠兔及高原松田鼠等野生动物, 这是狗捕食野生动物并与野生食肉动物竞争资源的直接证据。如果散养狗的种群数量不加以控制, 可能会持续增长, 通过捕食、竞争、排斥等方式对当地野生动物生存及生态系统稳定造成严重影响, 需要管理者及早加以重视并进行有效管理。
以往食性研究显示豹猫主要取食啮齿目动物, 其在豹猫粪便样品中的出现频率(%FO)可高达81%-96% (Austin et al, 2007; Rajaratnam et al, 2007; Shehzad et al, 2012a; Lee et al, 2014)。我们的分析结果中啮齿目也是豹猫食物中检出频率最高的类群(%FO = 100%), 但具体物种构成上与以往调查有所不同。泰国、巴基斯坦、韩国等地豹猫取食的啮齿目食物主要为大鼠属(Rattus)和小鼠属(Mus)物种(Austin et al, 2007; Shehzad et al, 2012a; Lee et al, 2014), 而我们在甘孜地区检测到豹猫食物中的啮齿类以姬鼠属、松田鼠属(Neodon)以及白腹鼠属为主, 与在四川北部平武地区豹猫食性研究结果类似(Xiong et al, 2017)。此外, 兔形目中高原鼠兔和高原兔出现频率最高(%FO = 83%-100%), 而此前仅在四川北部有豹猫取食兔形目物种的报道(Xiong et al, 2017)。豹猫的食物中还发现了家牦牛、毛冠鹿、野猪、麝(Moschus spp.)等大中型偶蹄类动物。由于DNA食性分析不能检测猎物的年龄, 无法得知具体取食的情况。但考虑到豹猫与这些偶蹄类动物成年个体体型存在较大差异, 直接捕食健康成体的可能性较小, 推测豹猫有可能捕食其幼仔或取食动物尸体。
豹和雪豹的样品较少, 均各1份, 分别只检出了豹的猎物中华斑羚和雪豹的猎物岩羊。有蹄类常是豹和雪豹食物的最优选择(Seidensticker, 1976; Shehzad et al, 2012b)。由于大中型有蹄类体型较大, 豹和雪豹一餐通常只摄入1种食物, 因此每份粪便中的食物物种数少, 符合其捕食行为特征(Shehzad et al, 2012b, 2015)。随着采样量的增加, 预期豹和雪豹被检出的食物多样性也会有所增加。
不同食肉动物有不同的食物选择, 可能体现其体型、生理、捕食策略等方面的差异。本研究食性分析的初步结果展示了当地7种食肉动物的部分主要食物, 但由于每种食肉动物采样量不足, 各食物物种在食肉动物食谱中所占比例可能不够准确, 后续需要更广泛、长期地收集食肉动物粪便样品, 以进行更加全面的食性研究, 从而深入了解食肉动物的食物网结构、食性选择、竞争关系和共存机制。
3.3 问题与展望
采用DNA宏条形码技术并借助生物信息学方法的高通量食性分析有着传统方法无可比拟的优势。它采用较短的条形码设计通用引物, 使降解的DNA更容易被扩增和检测, 降低了对样品质量的要求。由于测序通量及深度远高于传统测序方法, 高通量测序具有极高的灵敏度, 微量食物也更容易被检测出来。同时一次测序大量样品产生大量数据, 对于需要大规模样品分析的研究可以节省更多的时间和成本。由于形态学难以区分的物种通过DNA宏条形码技术也能被识别, 研究者可以快速、大量、精确地获得物种信息, 不需要预先长期积累物种分类学知识。
本研究结果表明, 基于粪便DNA和DNA条形码技术的物种鉴定和食性分析, 可以对研究地区的物种进行快速本底调查。对于本研究地区的食肉动物来说, 高寒高海拔的温带森林及高山草甸生态系统生产力低, 食物资源比较匮乏, 对有限食物资源的利用差异可能比时间和空间资源的分化对于物种共存更为重要。资源分配等生态数据的缺乏常常是高海拔地区食肉动物保护难以开展的重要原因(Wang et al, 2014)。从本次调查结果看, 川西高原地区分布有多种大型食肉动物, 精准分子食性分析可以从食物资源利用方面为该地区大型哺乳类食肉动物的共存机制提供科学数据, 从而更深入地了解当地生态系统结构和过程, 有助于更好地保护生态系统功能和物种多样性。
除了食性分析, DNA宏条形码的用途正迅速扩展到更多生态学研究领域。如用于植物多样性分析, 发现现有类群中的隐存分类单元等(Yoccoz et al, 2012)。Murray等(2013)通过DNA宏条形码技术调查了上千块来自澳洲西南的化石, 发现了一些当地物种, 还有灭绝或者是从未记录过的物种。Kelly等(2014)通过环境DNA评估海洋中的鱼类多样性及分布从而进行环境监测。在生物资源领域, DNA条形码可以帮助监控珍稀濒危物种的进出口贸易, 实现有害生物及入侵种的快速检验, 以及中药材市场管理和规范等(陈士林等, 2013)。此外, 日益更新的实验技术和数据分析方法更进一步推动了DNA宏条形码技术向更高效、量化和自动化的方向发展(Zhou et al, 2013; Shi et al, 2017; Zhang et al, 2017)。
尽管DNA条形码技术在近些年来飞速发展, 但仍存在缺陷与不足, 其应用潜能尚未完全开发。首先, 依赖DNA条形码的物种鉴定受到GenBank、EMBL、DDBJ等公共资源数据库收录情况的限制。在对一个地区进行分子物种鉴定时, 为避免由于各大公开数据库对地方特有种或隐存种序列收录不全面而导致无法分辨和准确鉴定物种, 需研究者先对该地区进行充分的本底物种调查并建立本地物种条形码数据库。其次, GenBank中收录的部分序列可能自身存在序列错误, 或序列对应物种信息错误, 因此仅依靠DNA序列比对进行物种鉴定也有误判风险, 需要研究者认真核实。再次, 单一DNA条形码对各类群物种的分辨率不同, 这可能会影响到物种鉴定的精确度。如本研究中, 12SV5条形码对雀形目物种分辨率较低, 同一条序列能与多个物种高度匹配, 只能将该MOTU归类为包含所有匹配物种的上一级分类单元。这类问题需要通过针对特定类群设计分辨度更高的条形码来解决。最后, 样品采集、DNA提取、PCR和高通量测序过程中均可能出现样品间的交叉污染问题, 导致假阳性结果。本研究采取多项措施减少交叉污染造成的假阳性问题: (1)在PCR扩增和测序中同时进行DNA提取空白对照及不加DNA模板的PCR空白对照反应来进行假阳性检验; (2)数据处理时删除每个样品中序列数少于该样品总序列数1%的MOTU以减少交叉污染影响; (3)对每个样品设置3次PCR平行对照反应, 在 ≥ 2次PCR中检测到某个食物MOTU时才认为结果有效。
总之, 随着DNA条形码和宏条形码技术被日益广泛地使用, 研究者需要在不断扩充共享数据库资源的同时加强对数据准确率的监管, 并在各类群条形码设计上不断优化和改善。同时需要强调的是, 对DNA条形码技术的推广不意味着可以忽视或取代传统分类学。对于分类尚不明确的物种, 需在传统分类学家的帮助下, 建立可靠的条形码数据库, 在此基础上, 生物条形码才能发挥越来越大的作用。
参考文献
Ecology and conservation of the leopard cat Prionailurus bengalensis and clouded leopard Neofelis nebulosa in Khao Yai National Park, Thailand
Terrestrial carnivores and human food production: Impact and management
DOI:10.1111/j.1365-2907.2008.00122.x URL [本文引用: 1]
Estimating population size of elusive animals with DNA from hunter-collected feces: Four methods for brown bears
Biodiversity loss and its impact on humanity
DOI:10.1038/nature11148 [本文引用: 1]
DNA barcoding of biological resources
生物资源的DNA条形码技术
Current progress of DNA barcoding
DNA条形码研究进展
OligoTag: A program for designing sets of tags for next-generation sequencing of multiplexed samples
DOI:10.1007/978-1-61779-870-2 URL [本文引用: 1]
Threats to ecosystems: Impacts and research needs
DOI:10.2307/2269562 URL [本文引用: 1]
Ultrasequencing of the meiofaunal biosphere: Practice, pitfalls and promises
DOI:10.1111/mec.2010.19.issue-s1 URL [本文引用: 1]
Feeding interactions in an assemblage of terrestrial carnivores in central Mexico
DOI:10.1186/s40555-014-0102-7 URL [本文引用: 1]
Diets of sympatric red foxes Vulpes vulpes and wild dogs Canis lupus in the Northern Rivers Region, New South Wales
DOI:10.1071/AM06013 URL [本文引用: 1]
Niche overlap between marsupial and eutherian carnivores: Does competition threaten the endangered spotted-tailed quoll?
DOI:10.1111/jpe.2008.45.issue-2 URL [本文引用: 1]
Biological identifications through DNA barcodes
DOI:10.1098/rspb.2002.2218 URL [本文引用: 1]
A global synthesis reveals biodiversity loss as a major driver of ecosystem change
DOI:10.1038/nature11118 [本文引用: 1]
Effects of biodiversity on ecosystem functioning: A consensus of current knowledge
DOI:10.1890/04-0922 URL [本文引用: 1]
Trophic relationships of sympatric small carnivores in fragmented landscapes of southern Brazil: Niche overlap and potential for competition
Using environmental DNA to census marine fishes in a large mesocosm
DOI:10.1371/journal.pone.0086175 URL [本文引用: 2]
High-throughput DNA sequencing concepts and limitations
DOI:10.1002/bies.200900181 URL [本文引用: 1]
DNA barcodes for ecology, evolution, and conservation
DOI:10.1016/j.tree.2014.10.008 URL [本文引用: 1]
Food habits of the leopard cat (Prionailurus bengalensis euptilurus) in Korea
DOI:10.3106/041.039.0107 URL [本文引用: 2]
Diet composition of wolves Canis lupus in the northeastern Qinghai-Tibet Plateau, China
DOI:10.1007/BF03194165 URL [本文引用: 1]
Evaluating the reliability of field identification and morphometric classifications for carnivore scats confirmed with genetic analysis
DOI:10.1002/wsb.549 URL [本文引用: 1]
Research progress in DNA minibarcoding and metabarcoding
DNA微型条形码和复合条形码研究进展
Food habits of Arctic wolves in Greenland
DOI:10.2307/1382859 URL [本文引用: 1]
Do wild dogs exclude foxes? Evidence for competition from dietary and spatial overlaps
DOI:10.1111/aec.2005.30.issue-5 URL [本文引用: 1]
Factors affecting the (in)accuracy of mammalian mesocarnivore scat identification in South- western Europe
DOI:10.1111/jzo.2013.289.issue-4 URL [本文引用: 1]
Food web complexity and higher-level ecosystem services
DOI:10.1046/j.1461-0248.2003.00469.x URL [本文引用: 1]
Scrapheap challenge: A novel bulk-bone metabarcoding method to investigate ancient DNA in faunal assemblages
DOI:10.1038/srep03371 [本文引用: 2]
Biodiversity hotspots for conservation priorities
DOI:10.1038/35002501 [本文引用: 1]
Carnivore conservation
DOI:10.2307/3803078 URL [本文引用: 1]
Who is eating what: Diet assessment using next generation sequencing
DOI:10.1111/j.1365-294X.2011.05403.x URL [本文引用: 4]
Ecometagenetics confirm high tropical rainforest nematode diversity
DOI:10.1111/mec.2010.19.issue-24 URL [本文引用: 1]
Diet and habitat selection of the leopard cat (Prionailurus bengalensis borneoensis) in an agricultural landscape in Sabah, Malaysian Borneo
DOI:10.1017/S0266467406003841 URL [本文引用: 1]
ecoPrimers: Inference of new DNA barcode markers from whole genome sequence analysis
DOI:10.1093/nar/gkr732 URL [本文引用: 1]
Status and ecological effects of the world’s largest carnivores
DOI:10.1126/science.1241484 URL [本文引用: 3]
On the ecological separation between tigers and leopards
DOI:10.2307/2989714 URL [本文引用: 1]
a) Carnivore diet analysis based on next-generation sequencing: Application to the leopard cat (Prionailurus bengalensis) in Pakistan
DOI:10.1111/j.1365-294X.2011.05424.x URL [本文引用: 4]
(
Forest without prey: Livestock sustain a leopard Panthera pardus population in Pakistan
DOI:10.1017/S0030605313001026 URL [本文引用: 1]
FuzzyID2: A software package for large dataset species identification via barcoding and metabarcoding using Hidden Markov Models and fuzzy set methods
Molecular identification of prey in predator diets
DOI:10.1046/j.1365-294X.2002.01471.x URL [本文引用: 1]
Towards next-generation biodiversity assessment using DNA metabarcoding
DOI:10.1111/j.1365-294X.2012.05470.x URL [本文引用: 1]
DNA barcoding for ecologists
DOI:10.1016/j.tree.2008.09.011 URL [本文引用: 3]
A new 2CTAB/PCI method improves DNA amplification success from faeces of Mediterranean (Barbary macaques) and tropical (lowland gorillas) primates
Blocking primers to enhance PCR amplification of rare sequences in mixed samples—A case study on prey DNA in Antarctic krill stomachs
DOI:10.1186/1742-9994-5-12 URL [本文引用: 1]
Environmental barcoding: A next- generation sequencing approach for biomonitoring applications using river benthos
DOI:10.1371/journal.pone.0017497 URL [本文引用: 1]
Dietary overlap of snow leopard and other carnivores in the Pamirs of Northwestern China
Feeding habits and niche partitioning in a predator guild composed of tigers, leopards and dholes in a temperate ecosystem in central Bhutan
DOI:10.1111/jzo.2009.277.issue-4 URL [本文引用: 1]
Progress on high-throughput sequencing and its applications in food web analysis
高通量测序及其在食物网解析中的应用进展
Molecular dietary analysis of two sympatric felids in the Mountains of Southwest China biodiversity hotspot and conservation implications
DOI:10.1038/srep41909 [本文引用: 5]
Molecular analysis of vertebrates and plants in scats of leopard cats (Prionailurus bengalensis) in southwest China
DOI:10.1093/jmammal/gyw061 URL [本文引用: 1]
Seasonal diet of wolves in the Dalaihu Nature Reserve, Inner Mongolia
内蒙古达赉湖自然保护区狼食性的季节性变化
Study methods of diets of carnivorous animals
食肉目动物食性研究方法
DNA from soil mirrors plant taxonomic and growth form diversity
DOI:10.1111/j.1365-294X.2012.05545.x URL [本文引用: 2]
BarcodingR: An integrated R package for species identification using DNA barcodes
DOI:10.1111/mee3.2017.8.issue-5 URL [本文引用: 1]
Food habits of wolf (Canis lupus) in Xing’anling Mountains
大、小兴安岭地区狼的食性
Description on foot tracts and fecal morphology of several carnivore species
几种食肉目动物的足迹与粪便形态描述
Review of non-DNA methods for identification of animal species
动物物种鉴定的非DNA方法评述
Ultra-deep sequencing enables high-fidelity recovery of biodiversity for bulk arthropod samples without PCR amplification
DOI:10.1186/2047-217X-2-4 URL [本文引用: 1]