鱼类多样性是水生生物多样性的重要组成, 是目前水生生态系统健康监测的关键指标之一(Zou et al, 2020)。传统的鱼类多样性监测方法多为网捕以及地笼等, 通过形态学鉴定和计数, 识别并记录其丰度(张春光等, 2011; Farriols et al, 2017)。但是传统方法存在生态破坏性大、稀有种和入侵种鉴别困难、耗时耗力、形态难以鉴定等缺点(徐念等, 2020; 王萌等, 2021; 沈梅等, 2022)。环境DNA宏条形码(environmental DNA metabarcoding)技术是从环境样本中捕获DNA并对其进行保存、提取, 使用针对目标类群的通用引物进行扩增, 结合高通量测序技术(high-throughput sequencing, HTS)及分类, 进而确定取样环境中多个目标生物分布状况的方法(Deiner et al, 2015; Thomsen & Willerslev, 2015)。该方法因程序化程度高、生态破坏性小以及检测灵敏度高, 作为传统方法的补充, 被广泛应用于生物多样性的监测(Civade et al, 2016; Yamamoto et al, 2017; Ruppert et al, 2019)。
目前, 已有诸多研究利用eDNA metabarcoding调查鱼类多样性(舒璐等, 2020; 徐念等, 2020; 王晨等, 2022), 如徐念等(2020)对长江中下游鱼类多样性的调查, 检测到20种鱼类, 比传统方法有更高的检测效率。也有学者设计并比较用于鱼类多样性调查的通用引物(Miya et al, 2015; Zhang et al, 2020), 优化eDNA采样方法和分析流程(Shaw et al, 2016; Schenekar et al, 2020)。但是国内运用eDNA metabarcoding调查鱼类多样性的研究相比国外较少(舒璐等, 2020; 徐念等, 2020, Zou et al, 2020), 更鲜有单独对清水鱼的讨论。清水鱼一般是指生活在清水中的鱼类, 和耐污鱼类不同, 清水鱼的生存对水质有一定的要求: 无污染且水源丰富(刘奕秋和方杰峰, 2003)。因此相对于其他鱼种, 清水鱼的分布更能够反映水质的优劣。
北京位于华北平原北部, 总面积16,410.54 km2, 山区面积约占总面积的62%, 地势为西北高、东南低。境内有永定河水系、潮白河水系、北运河水系、大清河水系、蓟运河水系五大水系。北京作为我国的首都, 人口密度大, 社会和经济发展迅速, 城市化率高, 这导致了鱼类栖息地的变化。杜龙飞等(2019)研究了北京市主要河流鱼类群落结构, 结果显示离城市越远的河流, 鱼类多样性越高。北京市鱼类多样性的调查研究相对较为全面, 但是调查时间比较久远, 较早的有张春霖(1933)的《中国鲤鱼志》和Wang (1936)的The Fishes of Peiping and Its Vicinity。其后, 北京自然博物馆王鸿媛(1994)的《北京鱼类和两栖、爬行动物志》以及陈卫等(2007)的《北京湿地生物多样性研究》。近些年, 张春光等(2011)调查北京及其邻近地区鱼类多样性, 共发现鱼类93种。
北京市虽然有较多的鱼类多样性调查结果, 但多是基于传统法。本研究则使用eDNA metabarcoding在2个季节对北京市北运河水系、潮白河水系以及大清河水系的鱼类多样性进行调查,筛选出清水鱼, 并分析其多样性及时空分布。旨在探讨环境DNA metabarcoding在监测北京市鱼类多样性及空间分布的可行性, 为北京市清水鱼生物多样性监测及保护提供新的技术手段。
1 材料与方法
1.1 环境DNA样本采集
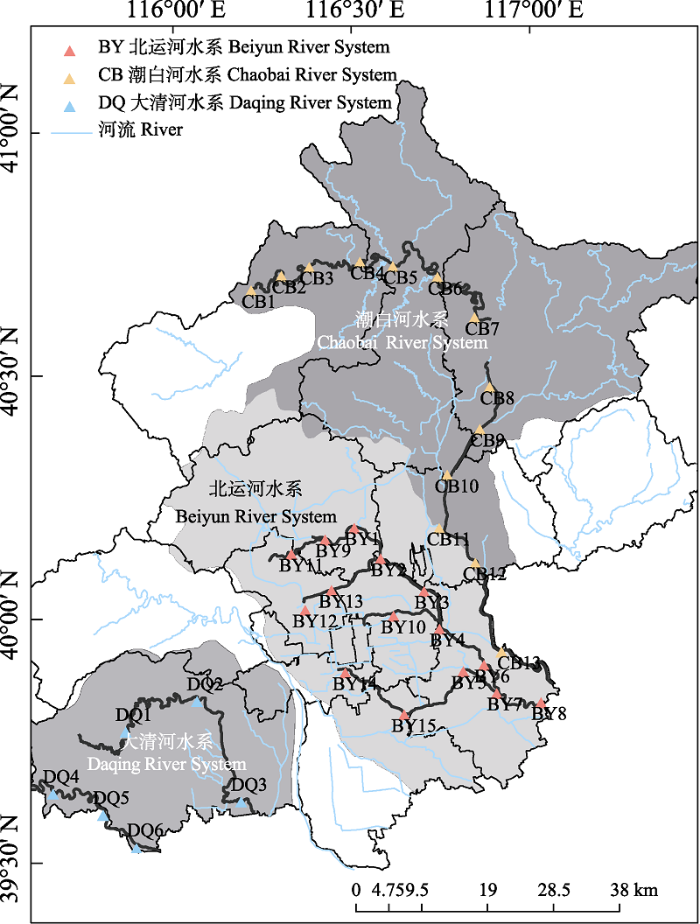
本次采样于2020年9-10月(秋季)和2021年6-7月(夏季)进行, 城区河流(北运河水系) 15个采样点和山区河流(潮白河和大清河水系) 19个采样点, 各采样点编号及分布见图1。使用容量为1 L的干净棕色塑料瓶采集河流岸边的表层水, 每个采样点3个重复, 以提高目标物种的检测概率(舒璐等, 2020; Zou et al, 2020)。34个采样点共采集102个样品, 两个季节为204个。所有的水样均在24 h内使用0.45 μm的尼龙滤膜进行真空抽滤。每次过滤的同时设置1个无菌水的阴性对照, 操作方法相同, 以评估是否存在外源DNA污染。抽滤后的滤膜放置在干净无菌的离心管中, 避光保存在‒20℃。采样过程中记录每个采样点的海拔(H)、水温(T)、溶解氧(DO)、电导率(EC)、总溶解固体(TDS)以及酸碱度(pH) 6个环境因子。
图1
图1
北京市主要河流采样点分布图
Fig. 1
Distribution map of sampling sites of major rivers in Beijing
1.2 eDNA提取
使用DNeasy Tissue and Blood DNA提取试剂盒 (Qiagen, Venlo, the Netherlands), 按照以下步骤, 完成水样的提取过程。首先将滤膜剪碎放置于2 mL的离心管中, 加入720 μL的ATL组织裂解缓冲液和80 μL的蛋白酶K; 再将样品放置56℃水浴锅中震荡3 h, 离心30 s (6,000 × g); 分别转移400 μL的解析液至2支2 mL离心管; 再加入400 μL无水酒精和400 μL AL裂解缓冲液的混合液, 漩涡5 s, 离心30 s (6,000 × g); 每个样品每次取600 μL于洗脱核上, 离心1 min (6,000 × g)后弃滤液, 洗脱柱和收集管重新组装, 重复四次后更换收集管; 向每个样品加入500 μL AW1洗涤缓冲液离心1 min (6,000 × g), 再加入500 μL AW2 洗涤缓冲液并离心2 min (13000 × g), 弃废液, 用1.5 μL离心管代替收集管; 最后向每个洗脱柱加入500 μL的AE洗脱缓冲液, 并放置于37 ℃水浴锅10 min后离心1 min (13,000 × g), 过滤到1.5 μL的离心管, 即为最终的DNA富集液。设置空白对照组, 所加入的试剂为样品组剂量的1/2, 其余操作与样品组一致。所得eDNA溶液快速置于‒20℃冷冻保存, 直至下一步PCR扩增。
1.3 目的基因片段扩增和高通量测序
每个样品进行3次重复扩增, 则每个点位有9次扩增, 阴性对照同样如此。使用鱼类通用引物MiFish-U (Miya et al, 2015)对eDNA模板进行PCR扩增, 扩增长度约163-185 bp。第1轮PCR的测定体系为30 μL, 包括15 μL的2 × Hieff® Robust PCR预混液, 1 μL的正向和反向引物, 10-20 ng DNA模板, 再加入ddH2O至30 μL。PCR反应程序为: 94℃预变性2 min, 30个循环包括: 98℃变性5 s, 50℃退火10 s, 72℃延伸10 s, 72℃最后延伸5 min。第2轮扩增引入Illumina桥式PCR兼容引物, 将其置于无菌PCR管中(200 μL)配制同第一步相同的反应体系, 遵循以下反应条件: 95℃预变性3min, 5个循环包括: 94℃变性20 s, 55℃退火20 s, 72℃延伸30 s, 72℃最后延伸5 min。
对PCR产物进行文库构建, 使用Illumina Miseq平台进行高通量测序(生工生物工程(上海)股份有限公司)。下机得到的双端序列数据(PE reads)首先根据overlap关系使用PEAR (Zhang et al, 2014) (v 0.9.8)将成对的reads拼接成一条序列, 然后按照条形码标签序列识别并区分样品得到各样本数据。
1.4 地笼捕获
本研究中使用的传统方法为地笼法, 34个采样点各放置3个长度为2 m的地笼, 地笼内放置一定的鱼饵, 地笼之间的距离为5 m, 放置时间为24 h, 以提高捕获到鱼类的概率。取水样与收地笼的位置及时间相同, 这样确保了2种方法之间所鉴定的鱼类组合的差异不是由于鱼类的洄游引起的。捕获后的鱼类经鱼类专家通过形态特征识别鉴定, 记录鱼的种类以及捕获条数。
1.5 数据分析
用FASTP (Chen et al, 2018)去除引物接头序列, 并对序列质量进行控制和过滤, 得到各样本的有效数据。再利用Usearch (v 11.0.667) (Edgar, 2010)优化各样本序列获得OTU代表序列, 选出与代表序列相似性在97%以上的序列, 生成OTU表格。通过使用BLASTN tool (Altschul et al, 1997)将OTU序列与MitoFish网站(
统计不同分类水平的物种数量、OTU数量和序列数量, 计算物种序列相对丰度。为了探讨不同采样点鱼类群落组成的差异, 本研究基于物种序列丰度统计的χ2相似性系数进行对应分析(correspondence analysis, CA), 使用R的vegan包完成。并使用以下公式计算不同水系鱼类群落的优势度指数Y (计算软件使用Excel):
其中, Ti为出现第i种鱼的样点数, T为总样点数, ni为第i种鱼类序列丰度, N为总序列丰度;
本研究基于各采样点的物种序列丰度计算α多样性, 并使用方差检验判断3条水系清水鱼多样性显著性差异, 以进一步比较清水鱼多样性在两个季节的分布是否存在差异, 使用软件Graphpad Prism (v 9.1.0)完成图形绘制。使用冗余分析(redundancy analysis, RDA)探究影响清水鱼群落分布的主要环境因子, 使用R的pacman包完成。Spearman相关性系数分析清水鱼丰富度与环境因子和灯光指数的相关性, 灯光指数的数据来源于2019年NPP- VIIRS合成年度数据(
2 结果与分析
2.1 物种组成
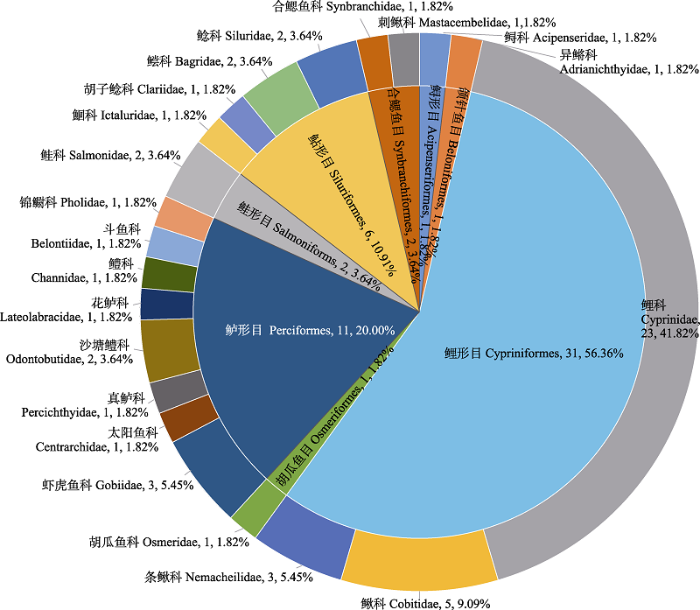
在本次调查中, eDNA metabarcoding共识别出鱼类8目21科47属55种。夏季46种, 隶属于6目16科38属, 秋季49种, 隶属于8目20科40属, 详见附录1。其中检测到的达氏鲟(Acipenser dabryanus)、团头鲂(Megalobrama amblycephala)、虹鳟(Oncorhynchus mykiss)、池沼公鱼(Hypomesus olidus)都是引入种或者人工养殖, 云鳚(Pholis nebulosa)和大口黑鲈(Micropterus salmoides)在咸淡水中生存。在目等级按照物种丰度占比从高到低排序为: 鲤形目(31, 56.36%)、鲈形目(11, 20.00%)、鲇形目(6, 10.91%)、合鳃目(2, 3.64%)、鲑形目(2, 3.64%)以及仅包含1种物种的颌针鱼目、鲟形目和胡瓜鱼目。其中又以鲤科(23, 41.82%)、鳅科(5, 9.09%)为主导, 物种分类情况可见图2。
图2
图2
eDNA metabarcoding识别鱼类物种在目和科分类学水平上的数量及所占比例
Fig. 2
Number and proportion of fish species identified by eDNA metabarcoding at the taxonomic level of order and family
2.2 物种时空分布
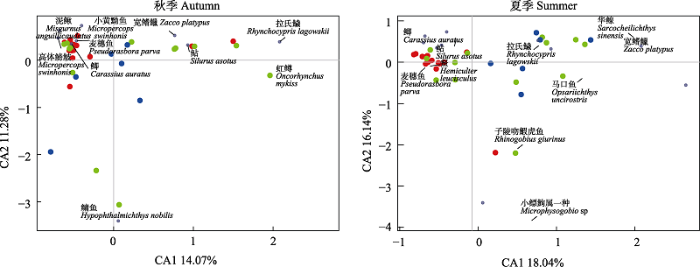
使用对应分析(CA)分别探究两个季节城区河流和郊区河流的物种组成(图3)。结果显示, 无论是夏季还是秋季, 北运河水系的样点之间较为集中,说明群落内物种差异不大。潮白河和大清河水系的样点分布较为分散, 群落内鱼类物种差异较大, 且和北运河水系之间的距离相对较远, 说明群落间物种组成存在差异。图3中标示的物种为序列丰度排名前十的物种, 由其分布结果可以看出, 鲫 (Carassius auratus)、麦穗鱼(Pseudorasbora parva)等距离北运河水系较近, 而清水鱼宽鳍鱲(Zacco platypus)、拉氏鱥(Rhynchocypris lagowskii)等距离潮白河和大清河较近。
图3
图3
3条水系在秋、夏季节鱼类群落组成分布。红色圆点表示北运河水系; 绿色圆点表示潮白河水系; 蓝色圆点表示大清河水系。
Fig. 3
Distribution of fish communities in the three river systems during the autumn and summer. Red dots represent Beiyun River System; Green dots represent Chaobai River System; Blue dots represent Daqing River System.
对3条水系的总体鱼类群落做优势种分析, 使用优势种指数Y进行表示。计算得到的优势种(Y > 0.02)结果见表1, 每条水系的物种按照优势度指数由上到下顺次排列。结果显示, 北运河水系优势种主要是一些耐污种类, 如鲫、麦穗鱼、泥鳅(Misgurnus anguillicaudatus)。而山区河流的优势种包含了华鳈(Sarcocheilichthys sinensis)、北鳅(Lefua costata)、宽鳍鱲、拉氏鱥等对于水质环境的要求较高清水鱼。
表1 3条水系鱼类群落优势种的优势度指数
Table 1
| 北运河水系 Beiyun River System | 潮白河水系 Chaobai River System | 大清河水系 Daqing River System | |||
|---|---|---|---|---|---|
| 物种 Species | 优势度指数 Dominance index (Y) | 物种 Species | 优势度指数 Dominance index (Y) | 物种 Species | 优势度指数 Dominance index (Y) |
| 鲫 Carassius auratus | 0.5175 | 鲫 Carassius auratus | 0.2556 | 宽鳍鱲 Zacco platypus | 0.1517 |
| 麦穗鱼 Pseudorasbora parva | 0.0888 | 拉氏鱥 Rhynchocypris lagowskii | 0.1031 | 鲫 Carassius auratus | 0.1475 |
| 小黄黝鱼 Micropercops swinhonis | 0.0453 | 宽鳍鱲 Zacco platypus | 0.0807 | 拉氏鱥 Rhynchocypris lagowskii | 0.0968 |
| 泥鳅 Misgurnus anguillicaudatus | 0.0448 | 鲇 Silurus asotus | 0.0474 | 小黄黝鱼 Micropercops swinhonis | 0.0540 |
| 高体鳑鲏 Rhodeus ocellatus | 0.0345 | 子陵吻鰕虎鱼 Rhinogobius giurinus | 0.0350 | 马口鱼 Opsariichthys uncirostris | 0.0408 |
| 䱗 Hemiculter leucisculus | 0.0205 | 麦穗鱼 Pseudorasbora parva | 0.0242 | 华鳈 Sarcocheilichthys sinensis | 0.0406 |
| - | - | - | - | 褐吻鰕虎鱼 Rhinogobius brunneus | 0.0400 |
| - | - | - | - | 子陵吻鰕虎鱼 Rhinogobius giurinus | 0.0376 |
| - | - | - | - | 鲇 Silurus asotus | 0.0340 |
| - | - | - | - | 北鳅 Lefua costata | 0.0241 |
| - | - | - | - | 鲤 Cyprinus carpio | 0.0233 |
2.3 清水鱼的组成及分布
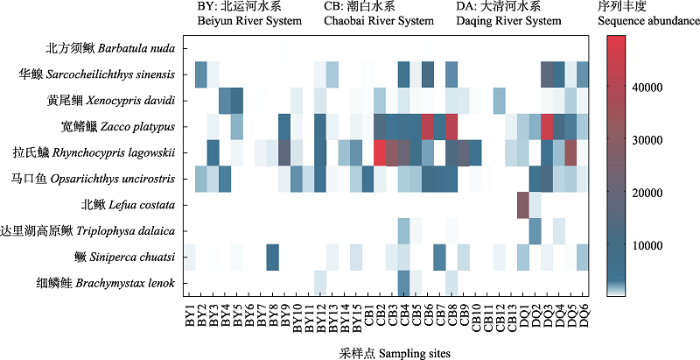
从总体鱼类结果中筛选出10种清水鱼, 其在每条水系的检测情况见表2。清水鱼中拉氏鱥(24)、马口鱼(24)、黄尾鲴(22)和宽鳍鱲(21)被检测到的点位最多。马口鱼主要分布在北运河水系和潮白河水系, 而黄尾鲴主要分布在潮白河水系。
表2 清水鱼在3条水系的检测的样点数情况
Table 2
| 物种 Species | 样点数 Number of detected site | |||
|---|---|---|---|---|
| 北运河水系 Beiyun River System | 潮白河水系 Chaobai River System | 大清河水系 Daqing River System | 总计 Total | |
| 北方须鳅 Barbatula nuda | 1 | 1 | 0 | 2 |
| 华鰁 Sarcocheilichthys sinensis | 4 | 5 | 4 | 13 |
| 黄尾鲴 Xenocypris davidi | 6 | 11 | 4 | 21 |
| 宽鳍鱲 Zacco platypus | 7 | 9 | 6 | 22 |
| 拉氏鱥 Rhynchocypris lagowskii | 9 | 9 | 6 | 24 |
| 马口鱼 Opsariichthys uncirostris | 10 | 9 | 5 | 24 |
| 北鳅 Lefua costata | 0 | 0 | 2 | 2 |
| 达里湖高原鳅 Triplophysa dalaica | 0 | 3 | 2 | 5 |
| 鳜 Siniperca chuatsi | 6 | 5 | 4 | 15 |
| 细鳞鲑 Brachymystax lenok | 1 | 3 | 0 | 4 |
图4
图4
34个点位清水鱼的序列丰度
Fig. 4
Sequence abundance of clearwater fish across 34 sampling sites
图5
图5
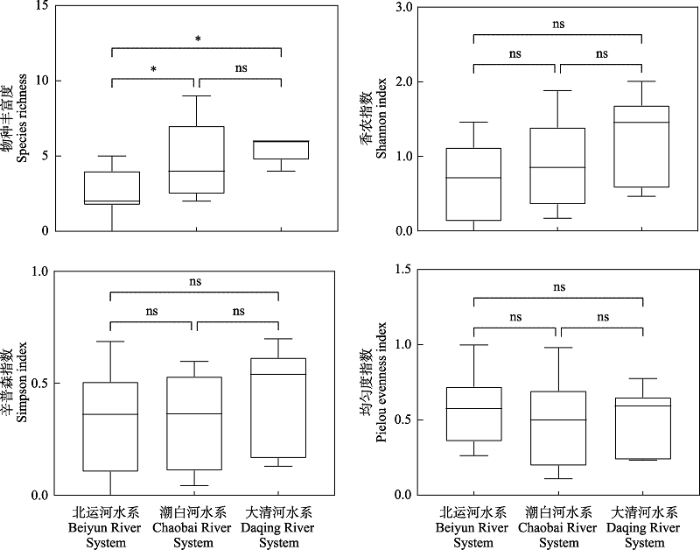
北京市3条水系清水鱼α多样性检验
Fig. 5
α diversity of clearwater fish in three river systems of Beijing. * indicates P < 0.05, ns indicates P > 0.05。
2.4 清水鱼多样性影响因素
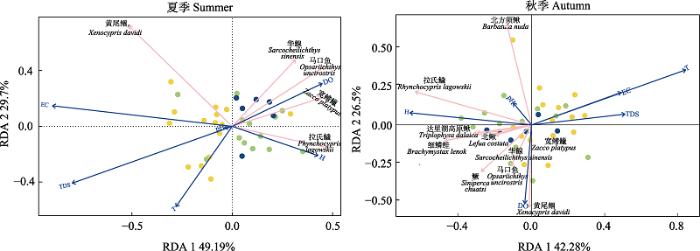
清水鱼群落组成受环境因子的影响, 并且在不同的季节的主要影响因子不同。附录2记录了本研究的所记录的环境因子数据。RDA分析(图6)表明前2个RDA轴分别解释了夏季和秋季鱼类群落结构差异的78.89%和68.78%。envfit检验显示总溶解固体(P = 0.0477)和电导率(P = 0.0127)对夏季清水鱼分布有显著影响, 城区水系和郊区水系之间有明显的区分; 但是秋季对清水鱼分布有显著影响的环境因子是温度(P = 0.0004)和海拔(P = 0.0428)。夏季清水鱼分布在高溶解氧的地方, 与电导率和总溶解固体呈负相关。秋季清水鱼分布与溶解氧和海拔呈正相关, 和温度呈负相关。
图6
图6
冗余分析(RDA)探究清水鱼群落结构与测量环境变量之间的关系。DO: 溶解氧; TDS: 总溶解固体; T: 温度; H: 海拔; EC: 电导率。黄色圆点表示北运河水系; 绿色圆点表示潮白河水系; 蓝色圆点表示大清河水系。
Fig. 6
RDA exploring the relationship between community structure and measured environmental variables. DO, Dissolved oxygen; TDS, Total dissolved solids; T, Temperature; H, Height; EC, Electrical conductivity。Yellow dots represent Beiyun River System; Green dots represent Chaobai River System; Blue dots represent Daqing River System.
图7
图7
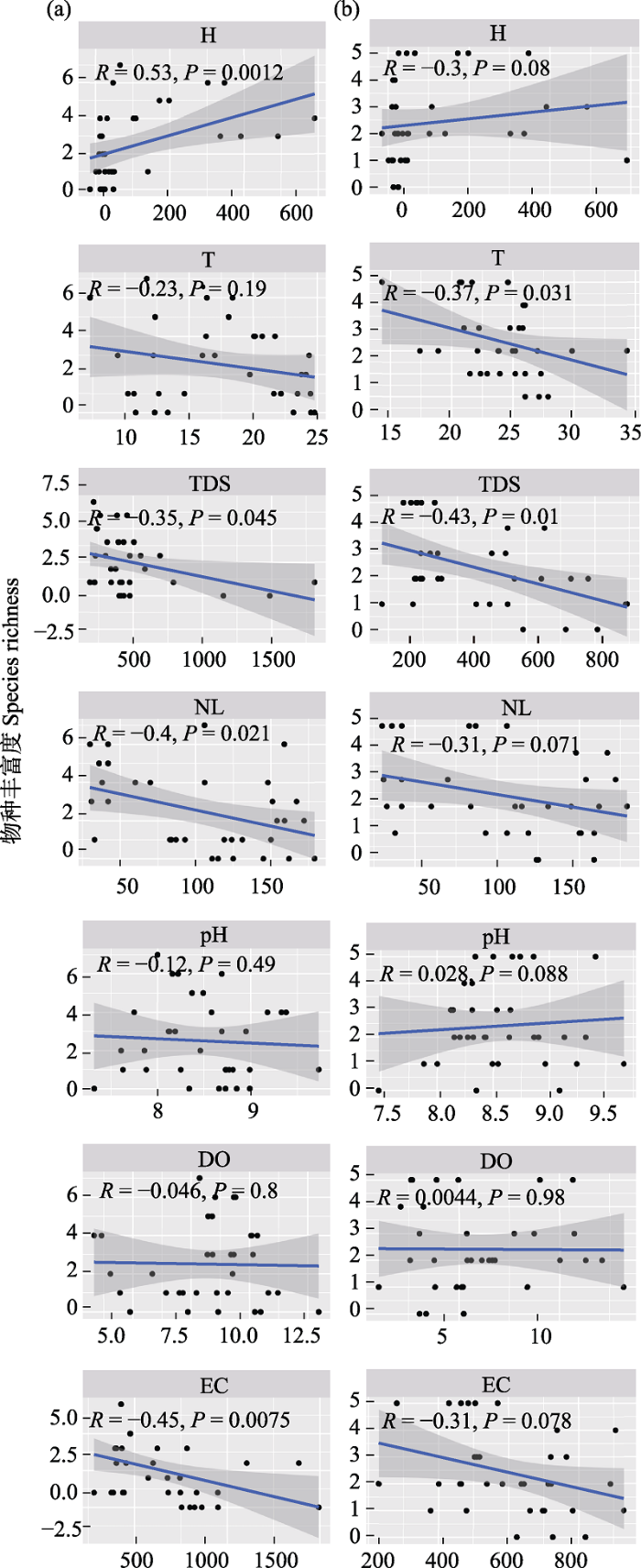
清水鱼丰富度与环境因子及灯光指数的相关性。H: 海拔; pH: 酸碱度; T: 温度;DO: 溶解氧; TDS: 总溶解固体; EC: 电导率; NL: 夜间灯光; a: 秋季; b: 夏季。
Fig. 7
Correlation of clearwater fish abundance with environmental factors and lighting indices. H, Height; pH, Potential of hydrogen; T, Temperature; DO, Dissolved oxygen; TDS, Total dissolved solids; EC, Electrical conductivity; NL, Night light; a, Autumn; b, Summer.
3 讨论
3.1 北京市主要河流鱼类多样性及分布
本研究使用eDNA metabarcoding检测出北京3大水系鱼类55种。其中以鲤形目鲤科居多, 总计23种, 占鱼类物种总数的41.82% (图2), 以鲫、麦穗鱼、小黄黝鱼、宽鳍鱲、拉氏鱥为优势种。附录1包含了地笼法所捕获到的鱼类种数, 共有35种, eDNA metabarcoding捕获的种数明显高于地笼法。但是2种方法获得的鱼类在大的分类单元组成结果相似, 同历史调查结果也一致(陈卫等, 2007; 张春光等, 2011; 杜龙飞等, 2019), 均表明北京市鱼类多样性以鲤形目和鲈形目为主。eDNA metabarcoding也检测到一些传统法及历史记录中没有的物种, 如达氏鲟、花䱻(Hemibarbus labeo)只在一些报道中出现过, 可能是属于人工养殖, 其DNA流入水体而被检测。同时也存在传统法捕获到, 但是eDNA metabarcoding没有识别到的物种, 如中华鳑鲏(Rhodeus lighti)、裸鲤(Gymnocypris przewalskii)、黑鳍鰁(Sarcocheilichthys nigripinnis)等, 推测是由于本研究没有分层采样, 而有些鱼类栖息在水体的底层, 如中华鳑鲏和黑鳍鰁, 因此在之后的研究中应做好分层采样, 增加检出概率。
北运河水系鱼类物种较潮白河水系和大清河水系更为均质, 点位间的物种组成相似性更高, 以鲫和泥鳅等耐污鱼类为优势种。作为北京市主要的纳污河, 北运河水系受人类干扰严重, 生境类型较为单一(张春光和赵亚辉, 2013), 其鱼类群落具有典型的城市受干扰河流鱼类特征。而潮白河水系和大清河水系点位间物种组成差异性较大, 以宽鳍鱲、拉氏鱥、北鳅等为优势种。潮白河水系和大清河水系, 多为山区河流, 生境类型相对自然, 水温较低,具有轻度干扰的山溪型鱼类特征(杜龙飞等, 2019)。这2种不同生境河流鱼类物种组成的差异和杜龙飞等(2019)使用传统法所得结果一致, 都表明了人类活动对北京市河流鱼类群落的影响。这说明eDNA metabarcoding能够用于调查北京市鱼类多样性, 且能展现鱼类群落空间格局。
3.2 北京市主要河流清水鱼的分布
3.3 清水鱼多样性及分布与环境因子的关系分析
生物的空间分布格局是环境异质性和人类干扰的长期影响的结果(Brown, 2000)。北京市清水鱼的分布在不同的季节中受不同环境因子的影响, 秋季主要受海拔和温度的影响, 夏季主要是电导率以及总溶解固体的影响。因为本研究条件有限, 测定的环境因子较少, 在以往的研究中, 发现有不同的环境因子影响着鱼类群落结构。Ji等(2022)和Kadye等(2008)认为水温是影响鱼类群落组成的重要因子, 王晨等(2022)发现鱼类群落影响因子有溶解氧等; 李捷等(2012)和Brown (2000)认为电导率和坡度是影响鱼类群落组成的主要环境因子; Xie等(2021)认为鱼类分布主要和营养元素有关; Smith等(2005)认为鱼类群落组成主要受海拔的影响, 但海拔是一个综合因素, 通常会直接影响温度与生境质量。Wu等(2020)研究渭河流域鱼类分布在夏季和秋季的影响因子发现河道宽度、pH值和流速对夏季鱼类分布有显著影响; 然而, 在秋季对鱼类分布影响显著的是pH值、总氮、总磷和流速。以上研究结果之所以有差异, 可能和研究生境以及时间有关。
本研究结果表明, 秋季清水鱼丰富度与电导率、总溶解固体以及夜间灯光指数呈显著负相关(P < 0.05), 但是与海拔呈显著正相关(P < 0.05)。夏季清水鱼丰富度和温度、总溶解固体呈显著负相关(P < 0.05)。总溶解固体显示了水体的污染程度, 无论是秋季还是夏季清水鱼多样性都随着水体污染程度增强而显著减少。夜间灯光指数显示了人类活动程度, 结果显示, 清水鱼多样性随着人类活动的增强而减少, 说明人类活动对清水鱼多样性消极的影响。
4 结论
本研究共得出以下结论:
(1) eDNA metabarcoding在北京市2个季节3条水系共识别出鱼类55种, 隶属于8目21科47属。鲤形目和鲈形目是调查区域的主要鱼类类群, 其中又以鲤形目鲤科为主, 占鱼类物种总数的41.82%。eDNA metabarcoding捕获率要高于传统法, 虽然无法代替传统法, 但是可作为快速调查北京市鱼类多样性的有效手段, 减少传统方法监测对北京水生生态系统的干扰。
(2)从空间分布上看, 城区河流鱼类组成更为均质化, 优势种以鲫、麦穗鱼、泥鳅等耐污鱼为主; 山区河流鱼类组成更为多样化, 以宽鳍鱲、拉氏鱥等鱼类为优势种, 物种组成的不同与2种生境环境因素及人为干扰有关。
(3)清水鱼在山区河流的多样性要显著高于城区河流。不同的季节影响清水鱼分布的环境因子不同, 夏季主要是总溶解固体以及电导率, 秋季主要是海拔和温度的影响。鱼类群落空间格局对人为活动的干扰具有显著的指示作用, 清水鱼多样性随着总溶解固体的升高以及人类活动的增强而显著降低。
附录 Supplementary Material
附录1 北京市主要河流分布鱼类名录
Appendix 1 Checklist of fish in major rivers in Beijing
https://www.biodiversity-science.net/fileup/PDF/2022240-1.pdf
附录2 本研究环境因子的测定
Appendix 2 Measurement of environmental factors of this study
https://www.biodiversity-science.net/fileup/PDF/2022240-2.pdf
参考文献
Gapped BLAST and PSI- BLAST: A new generation of protein database search programs
Fish communities and their associations with environmental variables, Lower San Joaquin River Drainage, California
DOI:10.1023/A:1007660914155 URL [本文引用: 2]
Fastp: An ultra-fast all-in-one FASTQ preprocessor
DOI:10.1093/bioinformatics/btx692 URL [本文引用: 1]
Spatial representativeness of environmental DNA metabarcoding signal for fish biodiversity assessment in a natural freshwater system
DOI:10.1371/journal.pone.0157366 URL [本文引用: 1]
Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA
DOI:10.1016/j.biocon.2014.11.018 URL [本文引用: 1]
Fish community characteristics and spatial pattern in major rivers of Beijing city
北京市主要河流鱼类群落的空间格局特征
Search and clustering orders of magnitude faster than BLAST
DOI:10.1093/bioinformatics/btq461 URL [本文引用: 1]
Bottom trawl impacts on Mediterranean demersal fish diversity: Not so obvious or are we too late
DOI:10.1016/j.csr.2016.11.011 URL [本文引用: 1]
Assessment of benthic invertebrate diversity and river ecological status along an urbanized gradient using environmental DNA metabarcoding and a traditional survey method
DOI:10.1016/j.scitotenv.2021.150587 URL [本文引用: 1]
Stream fish assemblages in relation to environmental factors on a montane plateau (Nyika Plateau, Malawi)
DOI:10.1007/s10641-008-9364-4 URL [本文引用: 1]
Relationship between fish community diversity and environmental factors in the Lianjiang River, Guangdong, China
DOI:10.5846/stxb201108041142 URL [本文引用: 1]
连江鱼类群落多样性及其与环境因子的关系
The methods of clearwater fish aquaculture in the mountains
山区清水鱼养殖方法
MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: Detection of more than 230 subtropical marine species
Past, present, and future perspectives of environmental DNA (eDNA) metabarcoding: A systematic review in methods, monitoring, and applications of global eDNA
DOI:10.1016/j.gecco.2019.e00547 URL [本文引用: 1]
Reference databases, primer choice, and assay sensitivity for environmental metabarcoding: Lessons learnt from a re-evaluation of an eDNA fish assessment in the Volga headwaters
DOI:10.1002/rra.3610 URL [本文引用: 1]
Comparison of environmental DNA metabarcoding and conventional fish survey methods in a River System
DOI:10.1016/j.biocon.2016.03.010 URL [本文引用: 1]
Methods and application of environmental dna detection of fish
环境DNA检测鱼类的方法及应用
Investigating the fish diversity in Erhai Lake based on enviromental DNA metabarcoding
基于环境DNA宏条形码的洱海鱼类多样性研究
Stream fish assemblages in relation to landscape position and local habitat variables
DOI:10.1577/T03-051.1 URL [本文引用: 1]
Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity
DOI:10.1016/j.biocon.2014.11.019 URL [本文引用: 1]
Research on the biodiversity of Qinhuai River based on environmental DNA metabarcoding
基于环境DNA宏条形码技术的秦淮河生物多样性探究
The Fishes of Peiping and Its Vicinity
Advances in the macrozoobenthos biodiversity monitoring and ecosystem assessment using environmental DNA metabarcoding
基于环境DNA宏条形码技术的底栖动物监测及水质评价研究进展
Assessment of aquatic ecological health based on determination of biological community variability of fish and macroinvertebrates in the Weihe River Basin, China
DOI:10.1016/j.jenvman.2020.110651 URL [本文引用: 1]
eDNA metabarcoding revealed differential structures of aquatic communities in a dynamic freshwater ecosystem shaped by habitat heterogeneity
DOI:10.1016/j.envres.2021.111602 URL [本文引用: 1]
Preliminary study on environmental DNA metabarcoding for detecting biodiversity in the middle and lower reaches of the Yangtze River
长江中下游环境DNA宏条形码生物多样性检测技术初步研究
Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea
DOI:10.1038/srep40368
PMID:28079122
[本文引用: 1]

Environmental DNA (eDNA) metabarcoding has emerged as a potentially powerful tool to assess aquatic community structures. However, the method has hitherto lacked field tests that evaluate its effectiveness and practical properties as a biodiversity monitoring tool. Here, we evaluated the ability of eDNA metabarcoding to reveal fish community structures in species-rich coastal waters. High-performance fish-universal primers and systematic spatial water sampling at 47 stations covering similar to 11 km(2) revealed the fish community structure at a species resolution. The eDNA metabarcoding based on a 6-h collection of water samples detected 128 fish species, of which 62.5% (40 species) were also observed by underwater visual censuses conducted over a 14-year period. This method also detected other local fishes (>= 23 species) that were not observed by the visual censuses. These eDNA metabarcoding features will enhance marine ecosystem-related research, and the method will potentially become a standard tool for surveying fish communities.
Fish species diversity and conservation in Beijing and adjacent areas
DOI:10.3724/SP.J.1003.2011.08072 URL [本文引用: 3]
北京及其邻近地区野生鱼类物种多样性及其资源保育
DOI:10.3724/SP.J.1003.2011.08072
[本文引用: 3]

作者2002–2010年间, 连续多年对北京及周边地区的野生鱼类进行了实地调查和采集, 结合对中国科学院动物研究所国家动物博物馆馆藏鱼类标本及相关文献资料的整理, 得出北京及其邻近地区分布过的鱼类计有93种, 隶属于13目23科73属; 去除引入种, 自然分布于该地区的原生野生鱼类为85种(包括经通海河流上溯至区域内的河口咸淡水、洄游性及原产但现为引入的物种), 隶属于12目21科65属。目前, 区域内自然—半自然水体中可以见到的野生鱼类计有49种(包括引入种6种), 原生的野生鱼类为43种, 隶属于6目12科36属。与历史记录相比, 从目到种各分类阶元的消失率均超过40%, 特别是原生野生鱼类的消失率更接近50%; 伴随着大量土著鱼类的消失, 物种组成的异质性明显降低, 物种多样性下降的程度十分明显。自然环境持续干旱、兴修水利、城市建成区面积迅速扩张和人口膨胀等引起的水域面积减小、原有水域环境的改变、过度捕捞等应是引起土著鱼类物种多样性急剧下降的主要原因。为了能使原有土著鱼类资源尽快得到更有效的保护和恢复, 建议进一步做好鱼类资源本底调查、进一步加强渔政管理包括对现有水生生物保护区的管理、筹建新的水生生物保护区、建立种质资源库、订立地方性水生野生动物保护名录、加强对水生野生动物保护的宣传教育、严控引入鱼种等。
PEAR: A fast and accurate Illumina Paired-End reAd mergeR
DOI:10.1093/bioinformatics/btt593 URL [本文引用: 1]
A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish
DOI:10.1111/2041-210X.13485 URL [本文引用: 1]
eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling
DOI:10.1016/j.scitotenv.2019.134704 URL [本文引用: 3]