1 引言
近一个世纪以来, 生物入侵、环境污染、气候变化等生态环境问题给全球生态系统带来巨大威胁。其中, 生物入侵已导致严重生态灾害, 成为五大全球性环境问题之一(鞠瑞亭等, 2012)。随着全球化进程的加快, 更加频繁的航运、水产品和宠物交易以及水产养殖引种等人类活动把长期地理隔离的物种带到同一水域, 导致外来水生生物在新的生态系统中快速传播, 引发生物入侵(Lockwood et al, 2013)。水生生物入侵由于隐蔽性强、所造成的灾害易扩散、治理手段缺乏等原因, 对入侵地生态系统具有很强的破坏性, 正严重威胁着水生生态系统的健康和稳定(Dudgeon et al, 2006; Hambler et al, 2011; Holland et al, 2012)。
我国国土面积广阔、海岸线长、港口众多, 沿海地区人口密度大、人类活动频繁, 加之长期实行的对外开放战略(建立自由贸易区和“一带一路”政策等), 为水生入侵生物的引入、定殖、扩散、灾害暴发等提供了便利条件, 成为受生物入侵影响最为严重的国家之一(Chen et al, 2017)。我国水生生态系统外来物种总数量已达553种, 包括429个淡水种和124个海水种, 其中66个入侵物种(淡水物种23个, 海水物种43个)已经引发了巨大的生态灾害(Chen et al, 2017)。入侵生物引发灾害的频率、程度和受灾面积在短时间内剧增, 严重破坏了入侵地生态环境, 改变了当地生物群落结构, 降低了入侵水域的生物多样性。其中, 最为典型的灾害事件即是由水生入侵藻类引发的赤潮。赤潮藻类生存能力极强, 是我国水生入侵物种中的第二大门类(目前共计发现15种入侵赤潮藻类), 这些藻类入侵后迅速成为区域优势种, 导致赤潮灾害频发(Chen et al, 2017)。据不完全统计, 我国每年由外来入侵生物造成的直接经济损失约2,000亿元人民币(万方浩等, 2009), 而其中由赤潮造成的经济损失高达5亿元人民币(王朝晖等, 2010)。
入侵生物一旦在水生生态系统中入侵成功, 将非常难以清除, 入侵生物的预防和治理已经成为相关领域科学家们关注的焦点。传统的治理手段包括物理法、化学法和生物法等(万方浩等, 2009; Lin et al, 2015; Chen et al, 2017)。然而, 这些方法在使用过程中都存在一定的局限性: 物理清除方法虽然简单方便, 但无法彻底根除入侵生物; 化学法见效较快, 但会使入侵生物产生耐药性, 并且容易造成水体环境的二次污染; 生物法作用温和, 但会一定程度上增加新物种入侵的风险(刘芳明等, 2007)。通过长期的实践探索发现, 以防为主、防治结合是最为有效的方式(Lin et al, 2015; Chen et al, 2017)。通过完善入侵生物档案库并构建早期监测预警技术体系和平台, 可对生物入侵的各阶段进行综合监控和管理, 并将危害损失控制在最低水平, 成为防控生物入侵的最直接有效的方式。与陆生生物相比, 水生生物的种类繁多、群落结构复杂, 很多生物类群具有形体微小、在入侵初期群体规模极小、隐匿于水下、可用于物种鉴定的外部形态缺乏等特点, 导致在水生生态系统中构建早期检/监测和预警体系在技术层面更具挑战(Xiong et al, 2016)。
由于入侵初期外来生物群体规模极小, 因此从复杂的群落中检测出这些低丰度生物便成为构建早期检/监测和预警体系的关键(Xiong et al, 2016)。水生群落中繁多的低丰度物种(生物量小于万分之一)是水生生态系统的重要组成部分。Sogin等(2006)提出“稀有生物区系(rare biosphere)”的概念, 即群落中所有稀有生物的集合。稀有生物区系主要包含两大类: 低丰度土著种(一部分为濒危种)和入侵早期的外来物种。低丰度土著种通常对环境胁迫更敏感, 在物质循环和能量传递中发挥着重要作用(杨江华, 2017; 陈义永等, 2018); 外来入侵种在引入初期虽然种群密度极低, 但随之而来的快速繁殖可使生态系统退化并加速本地种的灭绝进程(Pysek & Richardson, 2010)。
水生生物鉴定的传统手段依赖于显微镜或流式细胞技术(Shi et al, 2011; Weber & Pawlowski, 2013), 该方法工作量大, 无法满足快速大量监测的需求, 且灵敏度低, 难以监测和识别数量庞大的稀有生物区系(Galand et al, 2009; Cheung et al, 2010)。此外, 采样方式和物种自身形态特征也对水生生物物种鉴定工作产生了较大影响。水体的流动和季节性水文变化使取样的随机性大大增加(McDonald, 2004; Jerde et al, 2011)。样品保存不当会导致形态特征缺失, 给准确的分类鉴定造成困难(Briski et al, 2011; Darling & Mahon, 2011)。许多类群有变态发育的特点, 不同生活史阶段的形态结构和习性会发生很大变化; 部分类群在同一生活史时期的形态学特点极为相似, 也为物种鉴定带来了极大困难(Briski et al, 2011; Darling & Mahon, 2011)。上述这些因素直接影响了基于形态的水生入侵物种鉴定的准确度和效率, 因此亟需建立集快速精确、高灵敏度、高分辨率于一体的检测方法。环境DNA-宏条形码技术检测灵敏度高、耗时短、成本低、通量高, 成为鉴定外来水生入侵物种的强有力工具(Zhan & Maclsaac, 2015; Xiong et al, 2016), 在复杂群落稀有种的监测方面优势明显, 具有极大的应用前景(Zhan & Maclsaac, 2015; Xiong et al, 2016)。
本文系统总结了环境DNA-宏条形码技术的发展历程、基本特征及其在水生生物入侵早期监测与预警过程中的应用, 探讨了该技术应用的局限性并提出了可能的解决方案, 同时还对该技术在水生入侵物种监测和预警中的应用前景进行了展望, 期望为水生生物入侵的科学防控提供技术支撑。
2 环境DNA-宏条形码技术
2.1 环境DNA-宏条形码技术简介
环境DNA (environmental DNA, eDNA)是指直接从环境样品(例如土壤、空气、水体)中提取到的DNA, 是不同物种遗留在环境中的DNA的总和(Levy-Booth et al, 2007; Pietramellara et al, 2009)。水体中的环境DNA包含生物体经由皮肤、尿液、粪便、粘液等释放到环境中的DNA和细胞死亡裂解后释放到环境中的DNA (Taberlet et al, 2012; Rees et al, 2014)。
环境DNA-宏条形码技术主要操作流程为: 采集环境样本, 对样本中的环境DNA进行提取、基于分子标记基因设计引物对环境DNA进行PCR扩增及高通量测序, 将得到的大量序列直接与数据库中的序列进行比对, 或者先将序列聚类为可操作分类单元(operational taxonomic units, OTUs)后再进行比对, 达到同时对环境样本中多个物种(或高级分类单元)进行鉴定的目的。
2.2 环境DNA-宏条形码技术的关键环节
入侵物种监测包括对靶标和非靶标生物的监测。靶标物种的监测相对简单, 通过设计物种特异性引物监测靶标物种是否存在, 技术要点为保证引物的特异性、避免近缘或共存物种的错配造成非特异性扩增(郝雅宾等, 2018)。而对非靶标生物的监测对技术要求相对较高, 在选择高分辨率的分子标记基因的基础上, 设计高效率的通用引物, 通过分析群落组成和结构的动态变化从而实现早期监测与预警。检测稀有生物区系的组成是监测非靶标入侵物种的技术关键。基于环境DNA-宏条形码的监测体系目前的研究主要集中在两个方面: (1)分子标记基因的选择和通用引物的设计与评估; (2)高通量测序数据的处理及对关键参数的优化与评估。
2.2.1 高分辨率分子标记基因选择
选择适当的分子标记基因并设计高分辨率的通用引物, 是提高检测灵敏度和稀有物种检出率的关键。分子标记基因具有以下两方面特征: 有相对保守的区域用来设计通用性强的引物, 便于PCR扩增; 扩增区兼具种间差异大、种内差异小的特点, 能够有效区分不同物种(Zhan & Maclsaac, 2015; 吴彪等, 2018)。常见的真核生物分子标记基因包括线粒体细胞色素C氧化酶亚基I (COI)基因、内转录间隔区(ITS)、核糖体18S rRNA基因、28S rRNA基因和线粒体16S rRNA基因等。
不同分子标记基因适用范围不同, 需根据待检测生物类群以及研究目的综合选择。COI基因具有进化速度快和功能保守性强的特点, 常用来分析浮游动物、甲壳动物、微型后生动物等(Bourlat et al, 2013; Leray et al, 2013; Zhou et al, 2013; Avó et al, 2017)。但COI基因较快的进化速度给设计群落水平的通用引物带来很大困难(Zhan et al, 2014a)。海洋生态系统中的海绵动物、部分刺胞动物和水母类的线粒体DNA的进化速率比其他后生动物慢10-20倍, 进化速度不一致引起的DNA序列差异, 使得难以针对COI基因设计出能够将混合DNA样品中全部物种进行有效扩增的通用引物, 因而对海洋生态系统部分类群进行研究时不宜选用(Tang et al, 2012; Zhan et al, 2014a)。18S rRNA基因存在保守区和可变区且序列进化速度较慢, 适用于研究亲缘关系较远的生物(Brannock et al, 2016)。16S rRNA基因对刺胞动物具有较高的识别率(Bucklin et al, 2010)。Zhan等(2014a)比较了COI、16S rRNA和18S rRNA等分子标记基因在浮游动物环境DNA-宏条形码研究中的差别, 发现其所用的COI引物都无法提供高质量的PCR产物, 并建议使用18S rRNA基因和线粒体16S rRNA基因进行条形码研究以获得更多的物种信息。
2.2.2 高灵敏度通用引物设计
通用引物设计是环境DNA-宏条形码技术应用的基础, 也是对分析结果具有决定性影响的环节(Zhan & Maclsaac, 2015)。扩增引物的设计一般应遵从通用性强、灵敏度高等特点。通用性强意味着可以将复杂群落中的环境DNA进行有效扩增, 实现对绝大多数物种进行检测, 进而提高物种覆盖度(Zhan & Maclsaac, 2015); 高灵敏度通用引物可降低监测成本, 仅需进行一次PCR扩增即可完成对所有类群DNA片段的收集, 为检测复杂群落中低丰度物种提供了便利(Zhan et al, 2014a; Zhan & Maclsaac, 2015)。
高灵敏度引物的获得离不开对引物进行全面的评价和测试(Leray, 2013; Zhan et al, 2013)。引物测试需要基于从单一生物类群、复杂生物群落到稀有生物区系层层推进(Zhan et al, 2014a)。首先是对单一类群水平引物扩增稳定性和覆盖度的检测, 进而推广到复杂群落水平, 对引物进行覆盖度和PCR偏向性的检测。Leray等(2013)在研究天竺鲷科Nectamia savayensis、金鳞鱼科Myripristis berndti和Sargocentron microstoma的胃内容物时, 对COI基因设计了5组通用引物, 结果表明其中一对引物(mlCOIintF/jgHCO2198)比传统通用引物(LCO1490/ HCO2198)在后生动物中扩增效率更高, 能够获得更多的物种信息。最后是针对稀有生物区系的检测。目前较为常用的方法是内标法, 即通过在提取DNA之前加入群落中不存在的物种作为内标, 然后利用内标的绝对和相对丰度计算其他物种的绝对丰度。Zhan等(2014a)分析了淡水生态系统和海洋生态系统的典型群落, 成功设计出基于核糖体小亚基rDNA (SSU)的高分辨率通用引物, 通过内标法对基于通用引物Uni18S/Uni18SR新构建的宏条形码体系进行测试, 发现该体系灵敏度极高, 比已有方法提高了5个数量级。对复杂水生群落的PCR扩增结果显示, 此通用引物可以扩增出几乎所有水生真核生物类群(动物、原生生物、藻类、真菌等), 且扩增偏向性极小; 此通用引物将对低丰度入侵生物的检出率提高了5倍, 即仅存在一个幼虫的条件下, 检出率达到100% (Zhan et al, 2014a)。
2.2.3 重现性和检出率
重现性(reproducibility)指同一序列在各平行样本间同时出现的概率, 是衡量数据准确度和可靠性的重要指标(Prosser, 2010; Zhou et al, 2011)。Zhan等(2014c)对此问题进行了一系列研究, 通过对海洋和淡水两个生态系统浮游生物群落检测的结果进行比较分析发现, 只有39.8% (海洋)和36.4% (淡水)的OTU在几次重复中被同时检测到, 但与土壤微生物等其他复杂群落相比检出率已属较高(Zhan et al, 2014a)。研究同时发现, 低丰度OTU的重现性远低于高丰度OTU, 对于包含100条序列的OTU重现性可达到100%, 而单体(singletons, OTU中仅包含一个序列)的重现性则低于25% (Zhan et al, 2014a)。研究同时指出, 实验中各阶段的随机过程是重现性低的重要原因, 主要包括随机取样、DNA提取及PCR扩增的偏向性, 以及高通量测序深度不足和数据滤错(质量控制)环节过滤大量原始序列等(Zhan et al, 2014b, c; Zhan & Maclsaac, 2015)。
提高入侵种检出率是早期监测关注的核心问题。虽然针对高分辨率分子标记基因设计高灵敏度通用引物在一定程度上提高了检出率, 但尚有一些错综复杂的因素同时制约着检出率。目前已有通过内标和外标相结合的方法, 研究高通量测序及数据滤错环节对入侵种检出率的影响。结果表明, 数据滤错会影响稀有种检出率, 滤错严格度与检出率呈负相关关系, 造成此种结果的主要原因是低丰度生物对应的序列中存在测序质量不高等问题, 滤错过程将这些低丰度序列删除(Zhan et al, 2014b)。借助稀疏曲线(species rarefaction curve)可以更好地确定测序深度, 在一定范围内测序深度的增加会显著增加OTU检出数目, 特别是提高低丰度OTU的检出率。对序列丰度与检出率关系的研究表明, 虽然序列丰度与检出率总体呈正相关关系, 但对不同群落加入相同指示生物, 测序数据也并不能可靠地量化物种相对丰度(Sun et al, 2015)。近年来, 利用高通量测序技术结合无PCR扩增(PCR-free genome-skimming)的“超级条形码”技术, 可以解决由于PCR扩增偏向性造成的物种偏向性以及混合物种相对丰度难以分析等问题, 获得较准确的相对丰度信息, 有望以更高的灵敏度和准确性对混合物种样品进行定性及相对定量的监测(郎丹丹等, 2018)。增加生物学重复可以有效降低随机取样导致的误差, 同时, 结合实验重复(DNA提取、PCR扩增等)并增加测序深度, 谨慎处理原始数据和OTU聚类结果, 对于检出率的提高有极大的帮助(Zhan & Maclsaac, 2015)。
3 环境DNA-宏条形码技术在水生入侵物种早期监测和预警中的应用
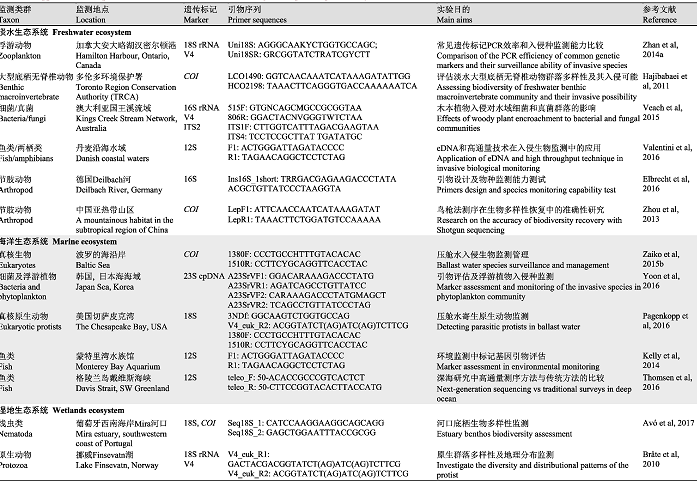
环境DNA-宏条形码技术以其高效、低成本的特点极大拓展了条形码技术在生物多样性分析、大范围物种检测以及入侵种监测等方面的应用(表1)。水生生态系统生物入侵的研究已从单一靶标生物的检出朝复杂非靶标物种、大尺度的方向发展, 在对靶标入侵种和非靶标入侵种的监测上都取得了良好的效果。
3.1 靶标入侵种的监测
入侵种检测的主要环节是针对靶标生物设计特异性引物, 通过检验环境DNA中是否含有目标种释放的DNA来判断是否存在靶标物种的入侵。Ficetola等(2008)基于美国牛蛙(Rana catesbeiana) Cyt-b基因设计特异性引物, 对法国18个自然水体的环境DNA样本进行扩增, 并在传统方法未检出的池塘中成功检测到该物种, 首次将环境DNA-宏条形码技术引入入侵物种监测领域。环境DNA与遥感、光谱等技术的联合使用为外来入侵物种的监测预警提供了更广阔的发展空间。Egan等(2015)结合现场准备光透射光谱(LTS)监测了压舱水中两种入侵贝类: 斑驴贻贝(Dreissena bugensis)和斑马贻贝(D. polymorpha)的入侵情况。目前, 这项技术被广泛应用于鲤鱼(Cyprinus carpio; Takahara et al, 2012)、新西兰泥螺(Potamopyrgus antipodarum; Goldberg et al, 2013)、克氏原螯虾(Procambarus clarkii; Tréguier et al, 2014)、鲢鱼(Hypophthalmichthys molitrix; Song et al, 2017)等入侵生物的监测, 监测物种类型涉及鱼、两栖、爬行、昆虫等多个门类。与国外相比, 我国环境DNA-宏条形码技术起步较晚, 目前仅在克氏原螯虾(马竹欣(2016)利用环境DNA技术调查入侵种克氏原螯虾在元阳梯田的分布. 硕士学位论文, 云南大学, 昆明.)、鱼类(姜维等, 2016; 徐念和常剑波, 2016; 郝雅宾等, 2018)等几个入侵物种的监测上有报道。随着环境DNA-宏条形码技术的不断发展, 其应用领域会逐步扩展到各种水生生态系统的多个物种(吴昀晟等, 2019)。
3.2 非靶标入侵种的监测
与靶标入侵种不同, 对非靶标入侵生物的监测需要设计高灵敏度通用引物, 通过比较群落物种组成变化达到监测预警的目的(单秀娟等, 2016)。Saunders (2005)通过对加拿大海区白蒙藻科的COI条形码设计通用引物, 发现了3个本来不存在于该海区的物种, 进而确定为入侵物种。Zaiko等(2015a)对航行不同时间的船舶压舱水进行采集, 然后利用环境DNA-宏条形码技术分析其中的物种数目, 探讨是否存在入侵种的可能。Thomsen等(2016)使用6对引物成功检测到15种入侵鱼类, 同时检测到4种入侵鸟类。Emily等(2016)对加拿大海岸线和五大湖主要港口的浮游生物样本进行分析后鉴定到379个浮游动物物种, 有24个物种被确定为外来入侵种, 其中11种为首次在该地区检测到。非靶标生物的监测不仅能够定性监测到入侵生物的存在, 还可以定量评估生物入侵程度(李飞龙等, 2018), 有利于监管部门针对入侵程度制定相应的治理措施, 成为早期预警系统的主要监测方式。
4 环境DNA-宏条形码技术存在的问题及优化方案
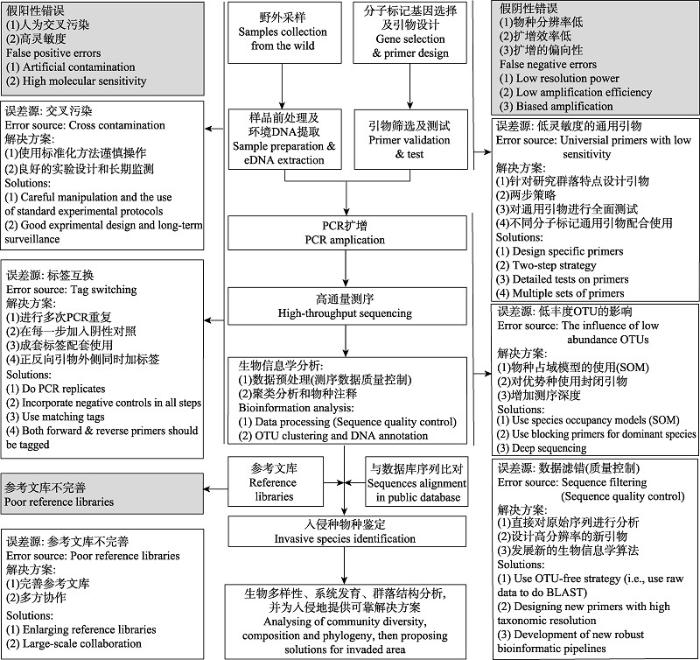
尽管环境DNA-宏条形码技术在水生入侵物种监测方面具有显著的优势, 但该技术在应用层面目前还存在一系列问题(图1)。我们对影响该技术监测效果的原因进行了讨论, 并为以后的研究提出可能的解决方案。
图1
图1
基于环境DNA-宏条形码技术的水生入侵生物早期监测与预警技术现存的主要问题及可能的解决方案
Fig. 1
A summary of error sources and possible solutions for both false positive and false negative errors when using environmental DNA-metabarcoding methods for early detection and warning of aquatic invasive species
4.1 环境DNA-宏条形码技术的准确性
环境DNA监测中缺乏灵敏度高的通用引物、监测准确度有待标准化方法的支撑、低丰度生物群落重现性较差、环境DNA产生和降解的动力学过程还不甚明确(Zhan & Maclsaac, 2015; 马鸿娟等, 2016)等问题会使监测中产生假阴性和假阳性两种结果, 从而影响监测结果的准确性。
4.1.1 环境DNA-宏条形码技术的假阴性
在实际监测过程中, 环境DNA-宏条形码技术存在I型和II型错误。I型错误是指拒绝实际上成立的正确假设, 为“弃真”的错误。环境DNA监测技术中的I型错误是指未能监测到环境中真实存在的物种, 降低检出率, 带来假阴性结果。I型错误为部分入侵种的定殖爆发成灾提供了时间, 对外来入侵种早期监测造成了严重的影响。分子标记片段分辨率低、通用引物扩增效率低、数据滤错时严格度设置过高以及PCR的偏向性是导致I型错误的直接原因。
序列分辨率的高低在很大程度上是由分子标记片段的选择造成的。低分辨率使得来源于不同物种的、具有高相似度的序列被分到同一个OTU, 可能使入侵物种被划为土著种。环境DNA-宏条形码鉴定物种的关键是寻找理想的分子标记基因。不同的基因片段在不同分类阶元系统进化研究中各有优缺点。已有研究表明, COI基因在鱼类不同属间的分辨率较低(Keskin & Atar, 2013; Deagle et al, 2014)。Zhan等(2014a)基于SSU基因研究了SSU V4-V9区域对入侵生物海鞘类群的分辨能力, 结果表明其分辨率在监测的类群间均可达到显著性水平。例如, 在玻璃海鞘属(Ciona)和二段海鞘属(Didemnum)的物种中种间差异大于5%, 但不同类群间的分辨率存在一定差异, 在拟菊海鞘属(Botrylloides)和菊海鞘属(Botryllus)等许多属中种间差异不到1% (Zhan et al, 2014a)。由于不同类群进化速度存在差异, 而目前大部分水生类群缺乏亲缘关系程度的研究, 故难以界定各物种间差异在何种水平会导致分子标记无法区分开的情况, 这就要求研究者在选择合适分子标记基因时, 对水生生物各类群的进化速度和差异程度有深入的了解, 并在通用性和分辨率两方面兼顾考虑。
当获取的环境DNA的量低于PCR能够检测到的阈值时, 目标物种将难以被检出。首先, 环境DNA在充满水和核酸酶的环境中极易随机降解成小片段, 目标片段的缺失会造成扩增困难。其次, 各类群在基因不同区域的多态性水平具有很大差异, 可能导致各类群的PCR过程中在引物结合位点发生不同程度的错配, 造成实际可以扩增的环境DNA总量低于检测阈值(Xiong et al, 2016)。高灵敏度的通用引物的使用可监测到生物总量占比仅2.3 × 10-5%的靶标物种, 尽管如此, 当入侵种生物量低于这个阈值范围时仍会出现假阴性结果(Zhan et al, 2014a)。
PCR的偏向性会导致低丰度和难扩增物种的数据丢失, 这是导致条形码解析度和普适性较低的重要原因(陈炼等, 2016)。PCR偏向性是由不同模板的扩增效率的固有差异(Polz & Cavanaugh, 1998), 或者是后期优势模板自我退火扩增的抑制作用(Suzuki & Giovannoni, 1996)造成的。同一体系中, 模板DNA间扩增效率的固有差异, 如GC含量高、富含二级结构使得不同物种对引物的亲和能力不同等均能导致PCR扩增效率的差异(Bellemain et al, 2010; Engelbrektson et al, 2010)。每轮PCR随机扩增都会带来偏向性, 使我们无法准确判断物种的原始相对丰度, 甚至发生漏检, 成为入侵种检出的障碍。
PCR扩增的偏向性可通过不断优化扩增条件(包括降低引物量、适当增加模板量、减少循环次数、适当提高退火温度)来加以改善。此外, 在明确自然条件下群落组成结构的情况下针对优势种设计封闭引物, 也可减少不必要的DNA扩增(Boessenkool et al, 2012)。同时, 可通过调整PCR程序, 例如采用巢式PCR方法提高扩增特异性(Davey et al, 2014); 或者使用两步策略: 在PCR第一轮扩增时, 基于18S rRNA等分子标记基因设计使用较为通用的引物, 对群落中的大多数类群进行扩增, 在第二轮专门为稀有类群设计特异性强的引物实现对低丰度类群的检测, 均可达到特异性富集的目的。
随着环境DNA-宏条形码技术的发展, 基于不同分子标记基因保守区设计的多对通用引物配套使用的多条形码技术可对群落进行更全面的监测(张宛宛 (2017) 基于DNA宏条形码技术的浮游植物群落多样性监测研究. 硕士学位论文, 南京大学, 南京.)。但多基因引物扩增得到的种类较为复杂, 给序列对比和多套数据整合等带来困难, 提高了假阳性率和监测成本。新一代的线粒体宏基因组技术, 即PCR-free技术(如DNA捕获法、鸟枪法测序等)摒弃了PCR扩增, 避免了PCR引入的一系列错误, 使结果更加精确。除了评估物种组成外, 通过推断每个物种中的线粒体DNA总量来评估各物种生物量乃至相对丰度, 是一种全新的方法。虽然PCR-free对生物信息算法提出了更高要求, 但该方法无疑为未来环境DNA-宏条形码技术提供了新的发展方向(Zhou et al, 2013)。
测序后一系列生物信息学分析方法的使用对检出率也存在极大影响。对原始数据进行数据滤错(质量控制)的过程中, 低质量序列的去除在一定程度上可避免“人为物种” (artifacts, PCR或测序错误导致的假序列)的产生, 但该过程又极易滤除环境中真实存在的稀有入侵种。真实条件下的稀有种和实验操作过程中产生的“人为种”的判断一直是困扰研究人员的问题。OTU聚类分析虽然能实现复杂水生生物群落的层级划分问题, 但会产生低丰度序列检出生物量低以及稀有生物区系检出困难的问题。在具体操作中, 对同一样本采用不同的聚类分析方法, 甚至同一聚类方法采用不同分类水平检出的入侵生物数目都有很大差异。有研究表明数量庞大的低丰度序列多为交叉污染造成, 对物种检出率影响很小(Kunin et al, 2010; Tedersoo et al, 2010)。但更多研究表明, 序列丰富度低的OTU如单体(singletons)、二体(doubletons)和三体(tripletons)是群落中真实存在的稀有种(Kauserud et al, 2012; Zhan et al, 2013)。这些问题的存在都对生物信息方法的选择与后期的数据处理分析提出了更高要求。
可以明确的是, 在入侵早期, 入侵物种处于低丰度水平, 因此低丰度物种的完整监测是不可忽略的。在序列比对环节, 将研究聚焦于比对平行样本间及各技术重复处理重现性高的序列和OTU可以提高低丰度物种的检出率。对于序列比对, 最为理想的方法是使用OTU-free的方法直接比对原始序列, 该方法在提出初期由于没有良好的算法导致工作量大、耗时长。随着环境DNA-宏条形码技术的不断发展, 层出不穷的新算法已经可以满足对原始数据的直接分析, 极大地降低了测序和算法导致的数据流失。一个有效的控制方法是在提高生物学重复的基础上使用物种占域模型(Species Occupancy Model, SOM; MacKenzie et al, 2002)评估监测结果。物种占域模型将空间占有率(occupancy rate)作为评估动物种群变化的一个度量标准(李勤等, 2013)。对入侵物种监测来说, SOM模型能估算出特定样点或区域被目标物种占有的概率, 预测目标地点、目标物种在未来的分布变化、种群动态趋势以及干扰程度对物种生存的影响(万雅琼等, 2017)。增加生物学重复能够进一步提高低丰度物种的检出率, Ficetola等(2015)建议根据样本特征确定重复水平, 对稀有分类群进行监测时采用8个以上的生物学重复, 重复次数的增多为SOM模型的准确性提供了保障。理论生态学模型仅需少量调查便能够通过重复测量数据更好地对实验中可能存在的问题进行补充指导, 对入侵物种的长期监测提供了很大的帮助。
4.1.2 环境DNA-宏条形码技术的假阳性
在统计学上II型错误是指错误地接受无效假设, 也就是犯了“存伪”的错误。在入侵种监测中, II型错误是指由于灵敏度高的高通量测序检测出不属于环境中的新序列并归为新群落, 凭空创造出“人为入侵种”带来假阳性结果, 造成人力物力的浪费。
交叉污染和分子标签互换是该现象出现的主要原因。有研究表明, 在监测的202个宏基因组中145个可能已发生污染, 占到总数的71.8%, 而每个宏基因组中污染序列所占比例更是高达64% (Schmieder & Edwards, 2011)。造成交叉污染的原因很多, 主要包括采样过程中采样器的污染以及各实验环节中样品间的相互污染等。PCR扩增时不可避免会形成异源双链(Kanagawa, 2003)、嵌合体(Odelberg et al, 1995)并产生随机性错误, 这些PCR虚假产物的检出是出现假阳性结果的原因之一。此外, 在分子操作过程中对大量平行样本进行标记后, PCR产物中可能会出现的标签互换(tag switching, 即由于合并样本带来的污染)现象也是交叉污染的间接来源。在早期的大量研究中, 为节约成本同时消除测序批次带来的系统误差, 在测序前处理时通常在引物外侧加入一段分子标签以达到对同一批次PCR扩增产物进行平行测序的目的, 通过标签对不同来源的序列进行区分, 进而分别进行序列拼接和数据处理及物种检出。这种操作存在一些潜在问题, 例如操作不当会导致存在少部分未连接上标签的引物序列, 保存不当会导致PCR产物稳定性下降使标签从引物上脱落或随机连接到其他序列上。这些情况都可能干扰扩增子测序, 导致序列读取错误并造成交叉污染, 发生II型错误(Carlsen et al, 2012)。
增加生物学样本重复数量是解决标签互换问题最为经济有效的方法。此外, 可以使用成套的标签同时对正向和反向引物进行标记, 避免相同批次样本对标签的重复使用(Carlsen et al, 2012)。每一步都设计相应的阴性对照, 既可以减少系统误差, 又可成为数据筛选时的重要参考。这种多因素导致的污染虽然难以避免, 但是缩短PCR产物的保存时间、严格遵循标准化方法并执行规范操作, 可以在一定程度上维持PCR产物的完整性并降低污染带来的影响。
4.2 物种参考数据库的完整性
利用环境DNA-宏条形码技术进行物种鉴定, 最终要回归到测序序列和参考数据库的比对上。因此, 参考数据库的完整性和质量直接决定了运用环境DNA-宏条形码技术进行物种鉴定的可靠性(杨江华, 2017)。虽然近年来参考数据库中物种信息增长很快, GenBank、Barcode of Life (BOLD)等公共数据库收录了大量数据, 但相对陆生生态系统, 对水生生态系统收录的类群较少。数据库的不完善使得大量的环境DNA-宏条形码数据不能得到正确注释, 从而大大降低了入侵种的检出率。Zhan等(2014a)的研究表明, 从加拿大安大略省汉密尔顿港收集的浮游生物群落样本通过高通量测序后获得的353个OTU中, 当以相似度99%拟合时, 只有32个(9.1%)与公共数据库中收录的可用序列相匹配。另一方面, 即使是收录在数据库中的物种, 由于分类学的错综复杂性, 也可能被归为不同的分类单元。此外, 文库中收录的数据多为COI和16S rRNA等常见分子标记基因的序列片段, 基于其他分子标记基因的数据较少, 在鉴定复杂群落和相似度较高的近缘种时不能满足需要。数据库收录的物种种类有限以及收录到的序列片段仅基于常见的分子标记基因, 使得文库数据在可用性上不尽人意。
随着环境DNA-宏条形码技术的高速发展, 世界各地在建的条形码数据库的开源与共享, 无疑为物种注释提供了丰富的资源。对于地方性入侵物种而言, 开展多方协作也是快速直接解决数据库问题的有效途径。
5 展望
生物入侵严重威胁着全球范围的水生生物多样性, 如果不及时采取有效的保护措施, 越来越多的本地物种将会以更快的速度面临濒危乃至灭绝。环境DNA-宏条形码技术具有高通量、高灵敏度、高分辨率的技术优势, 为水生生态系统入侵生物的早期监测预警系统的建立提供了条件。
随着测序技术的进一步发展, 第三代测序技术悄然而生, 单分子测序(如单分子实时测序技术、纳米孔单分子测序技术等)已逐渐成为新趋势。第三代测序技术在测序长度和准确性方面都有大幅度提高, 其极高的单分子分辨率及可以捕捉动态信息的优势是现有技术的补充。与第一代和第二代测序相比, 支持更高通量测序的第三代测序技术无需进行PCR扩增, 可以解决PCR扩增偏向性的问题, 使得序列比对分析将摆脱OTU聚类的方法, 这会极大程度上提高检出准确度。因此, 充分结合第三代测序技术优势, 优化环境DNA-宏条形码技术在水生入侵物种监测和预警过程中的应用, 是今后应该重点考虑的内容。
构建完善的物种DNA条形码的标准数据库及物种信息库、建立资源信息共享和应用平台是利用环境DNA-宏条形码技术进行入侵物种鉴定的基础。生物信息技术的发展将有助于环境DNA-宏条形码技术适应更加复杂多变的自然条件和物种类型, 提高监测灵敏度, 使得该项技术在更大的范围内得以推广应用(唐敏等, 2013)。环境DNA-宏条形码技术可以高效监测最易遭受生物入侵的区域, 在早期监测和预警中有着广阔的应用空间和发展潜能, 为水生入侵生物的检验检疫及立法工作提供数据基础和技术支持。该技术的发展将推动生物入侵早期监测进入一个崭新的时代, 为全球生物多样性保护做出卓越的贡献。
参考文献
DNA barcoding and morphological identification of benthic nematodes assemblages of estuarine intertidal sediments: Advances in molecular tools for biodiversity assessment
ITS as an environmental DNA barcode for fungi: An in silico approach reveals potential PCR biases
Metabarcoding reveals environmental factors influencing spatiotemporal variation in pelagic microeukaryotes
Freshwater Perkinsea and marine- freshwater colonizations revealed by Pyrosequencing and phylogeny of environmental rDNA
Use of DNA barcoding to detect invertebrate invasive species from diapausing eggs
Blocking human contaminant DNA during PCR allows amplification of rare mammal species from sedimentary ancient DNA
Genomics in marine monitoring: New opportunities for assessing marine health status
A “Rosetta Stone” for metazoan zooplankton: DNA barcode analysis of species diversity of the Sargasso Sea (Northwest Atlantic Ocean). Deep-Sea Research Part II: Topical Studies in Oceanography
Don’t make a mistake: Is tag switching an overlooked source of error in amplicon Pyrosequencing studies?
Application of environmental DNA metabarcoding in ecology
环境DNA metabarcoding及其在生态学研究中的应用
Biological invasions in aquatic ecosystems in China
Community structure of benthic macroinvertebrates and water quality assessment in the Songhua River
松花江流域大型底栖动物群落结构与水质评价
Composition and genetic diversity of picoeukaryotes in subtropical coastal waters as revealed by 454 Pyrosequencing
From molecules to management: Adopting DNA-based methods for monitoring biological invasions in aquatic environments
Forestry impacts on the hidden fungal biodiversity associated with bryophytes
DNA metabarcoding and the cytochrome C oxidase subunit I marker: Not a perfect match
Freshwater biodiversity: Importance, threats, status and conservation challenges
Detection of aquatic invaders using metabarcoding reveals a high number of non-indigenous species in Canadian ports
Rapid molecular detection of invasive species in ballast and harbor water by integrating environmental DNA and light transmission sectroscopy
Testing the potential of a ribosomal 16S marker for DNA metabarcoding of insects
Experimental factors affecting PCR-based estimates of microbial species richness and evenness
Species detection using environmental DNA from water samples
Replication levels, false presences and the estimation of the presence/absence from eDNA metabarcoding data
Unique archaeal assemblages in the Arctic Ocean unveiled by massively parallel tag sequencing
Environmental DNA as a new method for early detection of New Zealand mudsnails (Potamopyrgus antipodarum)
DOI:10.1899/13-046.1 URL [本文引用: 1]
Application of environmental DNA technology in the study of fish resources
环境DNA技术在鱼类资源研究中的应用
Environmental barcoding: A next-generation sequencing approach for biomonitoring applications using river benthos
Extinction rates, extinction-prone habitats, and indicator groups in Britain and at larger scales
Conservation priorities for freshwater biodiversity: The key biodiversity area approach refined and tested for continental Africa
‘‘Sight-unseen’’ detection of rare aquatic species using environmental DNA
DOI:10.1111/conl.2011.4.issue-2 URL [本文引用: 1]
Protocol optimization of eDNA analysis workflow for detecting Hucho bleekeri
以川陕哲罗鲑为目标物种的水样环境DNA分析流程的优化
Progress of biological invasions research in China over the last decade
近十年中国生物入侵研究进展
Bias and artifacts in multitemplate polymerase chain reactions (PCR)
High consistency between replicate 454 Pyrosequencing analyses of ectomycorrhizal plant root samples
Using environmental DNA to census marine fishes in a large mesocosm
DNA barcoding commercially important fish species of Turkey
Wrinkles in the rare biosphere: Pyrosequencing errors can lead to artificial inflation of diversity estimates
Qualitative and quantitative molecular construction of plant-pollinator network: Application and prospective
传粉网络构建的定性定量分子研究: 应用与展望
A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity: Application for characterizing coral reef fish gut contents
Cycling of extracellular DNA in the soil environment
Using environmental DNA metabarcoding to monitor the changes and health status of aquatic ecosystems
环境DNA宏条形码监测水生态系统变化与健康状态
Applications of camera trap in wildlife population ecology
相机陷阱在野生动物种群生态学中的应用
Introduction and use of non-native species for aquaculture in China: Status, risks and management solutions
Present situation, hazards and prevention and control measures of marine invasive alien species into China
中国外来海洋生物入侵的现状、危害及其防治对策
Invasion Ecology, 2nd edn. Blackwell Scientific Publications, Chichester
Estimating site occupancy rates when detection probabilities are less than one
Environmental DNA and its application in protecting aquatic ecosystem
环境DNA及其在水生生态系统保护中的应用
Template- switching during DNA synthesis by Therms aquaticus DNA polymerase
DOI:10.1093/nar/23.11.2049 URL [本文引用: 1]
Amplicon-based Pyrosequencing reveals high diversity of protistan parasites in ships’ ballast water: Implications for biogeography and infectious diseases
Extracellular DNA in soil and sediment: Fate and ecological relevance
Bias in template-to-product ratios in multitemplate PCR
Replicate or lie
DOI:10.1111/j.1462-2920.2010.02201.x URL [本文引用: 1]
Invasive species, environmental change and management, and health
The detection of aquatic animal species using environmental DNA: A review of eDNA as a survey tool in ecology
Applying DNA barcoding to red macroalgae: A preliminary appraisal holds promise for future applications
Fast identification and removal of sequence contamination from genomic and metagenomic datasets
Application of environmental DNA technology in aquatic ecosystem
环境DNA (eDNA)技术在水生生态系统中的应用研究进展
Plastid 16S rRNA gene diversity among eukaryotic picophytoplankton sorted by flow cytometry from the South Pacific Ocean
Microbial diversity in the deep sea and the underexplored “rare biosphere”. Proceedings of the National Academy of Sciences,
DOI:10.1073/pnas.0605127103 URL [本文引用: 1]
Making sense of the noise: The effect of hydrology on silver carp eDNA detection in the Chicago area waterway system
Unreliable quantitation of species abundance based on high- throughput sequencing data of zooplankton communities
Bias caused by template annealing in the amplification of mixtures of 16S rRNA genes by PCR
Environmental DNA
DOI:10.1111/j.1365-294X.2012.05542.x URL [本文引用: 1]
Kawabata Z (2012) Estimation of fish biomass using environmental DNA
The widely used small subunit 18S rDNA molecule greatly underestimates true diversity in biodiversity surveys of the meiofauna. Proceedings of the National Academy of Sciences,
The application of metabarcoding technology in identification of plant species diversity
Metabarcoding技术在植物鉴定和多样性研究中的应用
454 Pyrosequencing and Sanger sequencing of tropical mycorrhizal fungi provide similar results but reveal substantial methodological biases
Environmental DNA from seawater samples correlate with trawl catches of Subarctic, deepwater fishes
Environmental DNA surveillance for invertebrate species: Advantages and technical limitations to detect invasive crayfish (Procambarus clarkii) in freshwater ponds
DOI:10.1111/1365-2664.12262 URL [本文引用: 1]
Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding
Woody plant encroachment, and its removal, impact bacterial and fungal communities across stream and terrestrial habitats in a tallgrass prairie ecosystem
Site occupancy model: Principles and research progress
物种占域模型的基本原理及其研究进展
Control and management of harmful algal bloom species introduced by ballast water
船舶压舱水引起的有害赤潮藻类生态入侵及其控制管理
Can abundance of protists be inferred from sequence data: A case study of foraminifera
Comparative analysis of different DNA barcoding methods for Veneroida classification and identification
不同DNA条形码基因在帘蛤目贝类分类鉴定中的比较分析
The application of environmental DNA in the monitoring of the Yangtze finless porpoise, Neophocaena phocaenoides asaeorientalis
环境DNA在长江江豚监测中的应用
Early detection of invasive species in marine ecosystems using high-throughput sequencing: Technical challenges and possible solutions
Preliminary study on fish species detection in the middle and lower Yangtze River using environmental DNA
长江中下游干流环境DNA样本鱼类物种检测的初步研究
Adaptive Strategies of Clonal Plants Growing in Heterogeneous Environments
太湖流域浮游动物物种多样性与环境污染群落生态效应研究
.
Development of a cost-effective metabarcoding strategy for analysis of the marine phytoplankton community
Metabarcoding approach for the ballast water surveillance — An advantageous solution or an awkward challenge?
Metabarcoding approach for nonindigenous species surveillance in marine coastal waters
High sensitivity of 454 Pyrosequencing for detection of rare species in aquatic communities
a) Performance comparison of genetic markers for high-throughput sequencing-based biodiversity assessment in complex communities
b) Influence of artifact removal on rare species recovery in natural complex communities using high-throughput sequencing
c) Reproducibility of Pyrosequencing data for biodiversity assessment in complex communities
Rare biosphere exploration using high-throughput sequencing: Research progress and perspectives
Biological invasions in aquatic ecosystems in China
Reproducibility and quantitation of amplicon sequencing-based detection
Ultradeep sequencing enables high- fidelity recovery of biodiversity for bulk arthropod samples without PCR amplification
DOI:10.1186/2047-217X-2-4 URL [本文引用: 2]