

|
|
||
|
基于DNA宏条形码技术分析香螺食性
生物多样性
2025, 33 (1):
24403-.
DOI: 10.17520/biods.2024403
底栖动物是海洋生态系统的重要组成部分, 在调控海洋生态系统的物质循环和能量流动过程中扮演了关键角色。腹足类动物通过捕食与被捕食行为调控着底栖生态系统的稳定性, 研究腹足类生物的食性有助于我们理解这一调控过程。香螺(Neptunea cumingii)是我国北方海域一种重要的腹足类动物, 具备极高的生态价值和经济价值。但是, 我们对其食性组成及生态功能的了解并不透彻。因此, 有必要探明自然条件下香螺现场食物组成, 提升对腹足类动物在我国北方海域底栖生态系统中所起调控作用的认识。本研究借助4份野生香螺胃含物样品, 以18S rDNA V4区和V9区为标靶, 利用DNA宏条形码技术对其胃含物真核生物进行分析。结果显示, 在4个样品中, 18S rDNA V4区和V9区共分别获得265,161条和221,998条高质量序列, 分别占各自原始序列的93.16%和86.54%, 分别注释到141个和490个OTUs; 虽然18S rDNA V4区获得的优质序列数及占比均更高, 但其注释到的物种数比18S rDNA V9区偏少。两个可变区所有OTUs分属17个门类, 包括动物界10门、真菌界5门、植物界1门, 以及SAR超类群, 包括不等鞭毛生物(Stramenopiles)、囊泡虫(Alveolates)和有孔虫(Rhizaria); 在纲水平上, 腹足纲、辅鳍鱼纲、吸虫纲和色矛纲相对丰度在两个可变区中均排名前十; 在OTU水平上, 两个可变区中3个以上样品中均有检出的物种仅分别占5.67%和8.08%, 且其共同属于软体动物门、脊椎动物亚门、子囊菌门及SAR超类群。总体上, 18S rDNA V4区和V9区两段DNA条形码分析结果显示, 香螺胃含物中真核生物种类丰富, 包括动物(如环节动物、节肢动物、软体动物)、真菌、植物和原生生物, 其中最丰富的类群是腹足类、鱼类、吸虫和真菌。在多个样本中只检测到一小部分共有OTUs, 这表明不同香螺摄食种类多变。结果表明, 香螺现场食性是一种机会主义捕食者, 动物尸体及海底沉积物可能是香螺自然状态下主要食物来源, 但同时其具备一定的清理附着生物潜能及植食性能力, 饵料可驯化性较强。其饵料组成受生存微环境影响较大, 具备一定的饵料驯化潜力。研究结果为深入了解香螺在海洋生态系统中的作用提供了数据支持, 并为香螺人工养殖饵料配比研究提供了新见解。 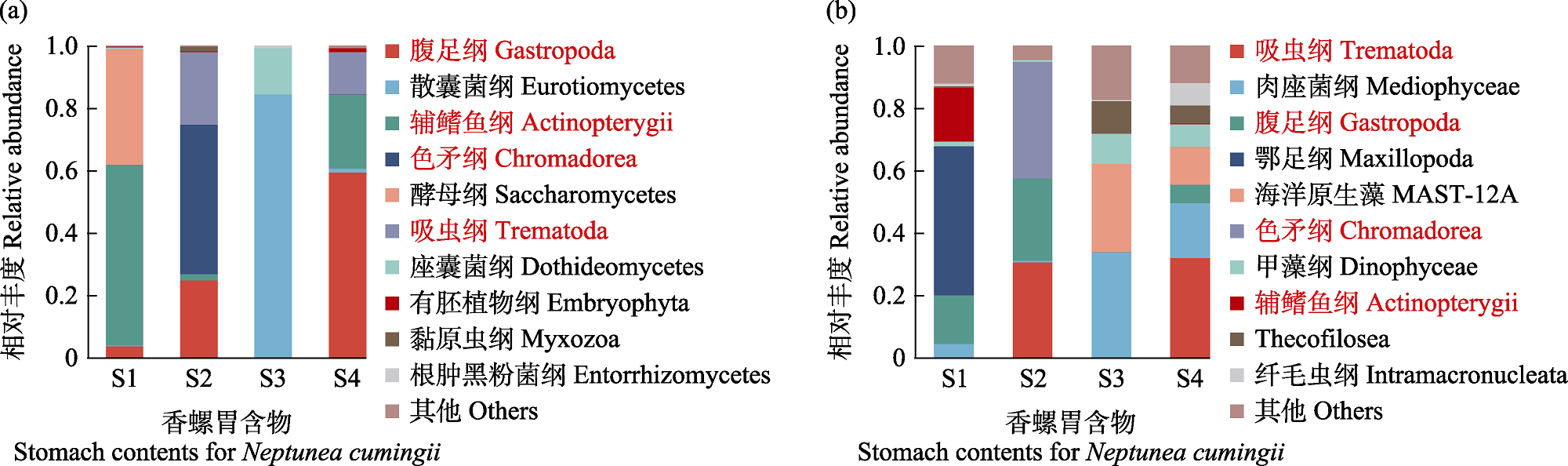 View image in article
图2
香螺胃含物纲水平相对丰度组成(S1‒4代表不同样品)。(a) 18S rDNA V4区; (b) 18S rDNA V9区。图例中红色字体为18S rDNA V4和V9区共同检出类别。
正文中引用本图/表的段落
利用18S V4区作为鉴定手段, 在香螺胃含物中序列相对丰度排名前10位的纲分别是腹足纲(21.92%)、散囊菌纲(21.48%)、辅鳍鱼纲(20.93%)、色矛纲(12.01%)、酵母纲(9.29%)、吸虫纲(9.25%)、座囊菌纲(3.82%)、有胚植物纲(0.42%)、黏原虫纲(0.40%)、根肿黑粉菌纲(0.18%)等(图2a); 利用18S V9区作为鉴定手段, 香螺胃含物中序列相对丰度排名前10位的纲分别是吸虫纲(15.63%)、肉座菌纲(14.04%)、腹足纲(11.98%)、鄂足纲(11.95%)、MAST-12A (10.17%)、色矛纲(9.34%)、甲藻纲(4.79%)、辅鳍鱼纲(4.37%)、Thecofilosea (4.21%)、纤毛虫纲(2.07%)等(图2b); 其中, 腹足纲、辅鳍鱼纲和吸虫纲在两组引物鉴定结果中均排名前10位, 表明其可能为香螺自然界中主要的食物来源或胃内寄生虫; 散囊菌纲、酵母纲、座囊菌纲及肉座菌纲虽未在两组引物中均为相对丰度排名前10位的物种序列, 但均隶属于子囊菌门, 表明子囊菌门可能为自然条件下香螺食物中重要的真菌来源; 同样, 黏原虫纲、MAST-12A、Thecofilosea及纤毛虫纲等虽未在两组引物中均被鉴定为相对丰度排名前十的物种序列, 但其均同属SAR超类群, 表明SAR超类群可能为自然条件下香螺重要的食物来源。
运用UPARSE软件(Stackebrandt & Goebel, Freshwater Perkinsea and marine-freshwater colonizations revealed by pyrosequencing and phylogeny of environmental rDNA 1 2010 ... PCR primers for species identification for stomach contents of Neptunea cumingii based on different hypervariable regions of 18S rDNA
本文的其它图/表
|