理解种群遗传结构和形成机制对于物种保护非常重要(Manel et al, 2003), 当物种内种群间具有较高的遗传分化且扩散受限制时, 繁殖群体应对不确定的局部环境变化的能力会下降(Walther et al, 2002)。划定繁殖群体精确的地理界线能够更准确地对种群数量动态进行监测, 从而提出更有效的管理计划(Palsboll et al, 2007; Funk et al, 2012)。
基于种群遗传学方法划分繁殖种群并评估种群连续性是制定有效保护策略的重要依据(Funk et al, 2012)。阿德利企鹅(Pygoscelis adeliae)是南极陆地动物的代表类群, 约占南极鸟类生物量的90% (Croxall & Prince, 1979; Borowicz et al, 2018)。阿德利企鹅的繁殖地点集中在南极大陆无冰的沿海岸区域, 常形成高密度的繁殖地, 目前最大的繁殖地约有75万个繁殖对(Borowicz et al, 2018)。冬季在环南极大陆的海冰上生活, 夏季初期到达南极大陆沿海岸的无冰区域结成繁殖对, 个体表现出强烈的归巢行为(Ainley et al, 1983), 这种行为可能是一种隔离机制, 对基因流形成阻碍。利用线粒体DNA以及少量核基因对西南极大陆阿德利企鹅的研究(Gorman et al, 2017), 以及利用单核苷酸多态性位点(single nucleotide polymorphisms, SNPs)对泛南极区域的研究(Clucas et al, 2018), 尚未发现各繁殖群体间存在显著的遗传分化。然而这些研究都缺少罗斯海地区阿德利企鹅的样本。作为冰川时代阿德利企鹅的重要避难所, 罗斯海区域的阿德利企鹅种群可能具有独特的遗传特征(Ritchie et al, 2004)。Shepherd等(2005)利用微卫星分子标记对恩克斯堡岛(74°53° S, 163°45° E)现生阿德利企鹅与古阿德利企鹅的比较研究发现, 现生种群与古种群之间存在明显的遗传分化, 但未对现生各繁殖种群之间的遗传分化程度进行分析。
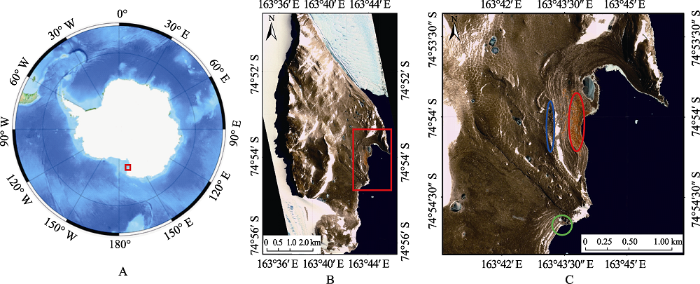
我国拟在南极罗斯海地区的恩克斯堡岛建立第五个南极科学考察站。出于环境保护、科学研究和发展战略的需要, 目前由中国牵头, 意大利和韩国参加, 在恩克斯堡岛北部、距离站址3 km处建立南极特别保护区。在该区域共有阿德利企鹅25,013个繁殖对(2018年1月韩国考察队调查数据, Hosung Chung, 个人通讯), 由花岗岩形成的陆岬将该区域划分为海景湾和南湾两个繁殖区域, 其中海景湾区域分布有24,940个繁殖对, 南湾区域分布有73个繁殖对。对挖掘出来的恩克斯堡岛的古阿德利企鹅骨骼、蛋壳、粪便, 通过碳稳定同位素进行年代判定, 发现阿德利企鹅利用海景湾与南湾持续作为繁殖地的历史超过7,000年(Baroni & Orombelli, 1991), 也是罗斯海地区全新世以来持续繁殖时间最长的种群(Baroni & Hall, 2004; Emslie et al, 2007; Lorenzini et al, 2010)。保护区内阿德利企鹅繁殖群自1963年就有监测记录(Stonehouse, 1969), 是迄今罗斯海地区阿德利企鹅最早的研究记录(Woehler & Croxall, 1997), 并一直持续到现在(Lyver et al, 2014), 对于研究气候变化对种群动态的影响有重要的价值。
阿德利企鹅具有较强的归巢行为, 基于少量个体的标记观测结果发现, 罗斯海地区的阿德利企鹅的归巢行为会受到冰山位置变动的影响(Shepherd et al, 2005), 但由于遗传数据的缺乏, 也还无法评估归巢行为的波动是否会对这一地区不同繁殖群体之间的基因流造成影响, 因此目前难以确定是把恩克斯堡岛的阿德利企鹅划分为一个大的保护单元还是几个小的保护单元。此外, 海景湾繁殖地存在常年积雪的带状地区(海拔20 m, 图1), 而阿德利企鹅仅在裸露的地面繁殖, 雪线对该区域阿德利企鹅繁殖群的隔离是否导致遗传分化, 也是划分保护单元的重要影响因素。
图1
图1
阿德利企鹅采样区地图。A: 南极地图, 红框示恩克斯堡岛的位置; B: 恩克斯堡岛地图, 红框示拟建保护区的位置; C: 采样区域, 蓝色和红色圆圈分别为海景湾高海拔和低海拔采样区, 绿色圆圈为南湾采样区。
Fig. 1
Sampling sites of Adélie penguins (Pygoscelis adeliae) in Inexpressible Island. (A) Map of Antarctic (Red rectangle: Inexpressible Island). (B) Map of Inexpressible Island (Red rectangle: the proposed Antarctic Specially Protected Area). (C) Sampling areas in Inexpressible Island. Blue circle, high altitude area in Seaview Bay; red circle, low altitude area in Seaview Bay; green circle, South Bay.
中国主持完成的设立保护区的预评估报告已经在第41次南极条约体系协商会议上得到通过, 并同意由中方牵头撰写保护区管理计划草案, 该草案已经过美国、意大利、新西兰、韩国和德国科学家的多轮讨论, 但大家在边界确定上还存在分歧。南湾的阿德利企鹅种群是否为特殊的小种群, 这涉及到与繁殖地相接海域(也是我国科考船在科考站后勤保障中可能停靠的区域)的保护范围。加强对恩克斯堡岛阿德利企鹅的研究和保护, 不仅可以提升我国对南极鸟类研究的水平, 也可以体现我国环境保护和参与全球环境治理的理念和行动。我们采集了恩克斯堡岛阿德利企鹅海景湾和南湾两个主要繁殖区域的样品, 利用基因组重测序的方法筛选单核苷酸多态性位点进行遗传多样性分析, 以检测不同地点的群体是否具有显著的遗传分化, 明晰种群结构, 从而划定重点保护种群, 为制订更为有效的保护计划提供信息, 同时也为保护区边界的划定提供理论依据。
1 材料与方法
1.1 研究区域
恩克斯堡岛位于南极罗斯海(74°54° S, 163°39° E), 阿德利企鹅繁殖地集中于恩克斯堡岛北部的海景湾和南湾区域。企鹅的栖息地沿冰川漂砾堆积的冰碛陇, 形成不同高度的14级阶地地貌, 区内海滩在全新世抬升高度达30 m (Baroni & Orombelli, 1991), 冰碛陇南北走向, 西高东低, 总体形成西北突出的弧形, 企鹅的巢见于不同级阶地中。
区内气候条件严酷, 依据1988-2012年气象资料, 年平均气温为-18.5℃, 每年低于-40℃的天数不超过0.1%, 年均风速12.0 m/s, 日均最大风速为34.2 m/s, 最大瞬时风速45 m/s。在持续的强下降风及Drygalski冰舌对浮冰阻隔的作用下, 当周围其他水域都覆盖固定冰或浮冰的时候, 在海景湾和南湾附近的特拉诺瓦湾形成了面积约为1,300 km²的巨大冰间湖(Bromwich & Kurtz, 1984), 为阿德利企鹅繁殖期觅食育雏提供了条件, 这也可能是这些阿德利企鹅长期在此繁衍的主要原因(Emslie et al, 2007)。
1.2 样品采集与基因组DNA提取
2018年1月参加我国第35次南极科学考察期间, 我们在恩克斯堡岛共采集自然死亡的阿德利企鹅样品147个, 采集样点见图1, 其中在南湾区域采集样品15个, 在海景湾高海拔区域(海拔20-58 m)采集样品44个, 低海拔区域(海拔1-20 m)采集样品88个。样品置于无水乙醇-20℃保存。
使用TIANamp Genomic DNA Kit血液/组织/细胞基因组DNA提取试剂盒(离心柱型)(天根, 北京)提取基因组DNA。使用Qubit 3.0荧光定量仪测量DNA浓度, 并利用琼脂糖凝胶电泳检测基因组DNA的完整度。将浓度较高且基因组DNA比较完整的86个样品进行全基因组重测序分析。首先通过Covaris系统将基因组随机打断成长度为350 bp左右的片段, 采用TruSeq Library Construction Kit (Illumina, 美国)进行建库, 在Illumina HiSeq平台以PE 2×150 (Paired-End 150 bp)模式进行测序。
1.3 数据分析
1.3.1 原始测序数据过滤和比对
将原始测序序列(raw reads)进行过滤, 去除接头和低质量的reads, 得到clean reads, 进行后续分析。采用阿德利企鹅De novo基因组作为参考基因组(Zhang et al, 2014), 应用BWA (Li & Durbin, 2010)将重测序结果比对至参考基因组。
1.3.2 SNPs检测及筛选
利用Sentieon DNAseq 软件包v.201711.05 (Freed et al, 2017)获取SNPs位点(emit_conf = 10, call_conf = 30)。应用Perl v5.10.1编写脚本, 保留在个体平均测序深度的1/3至2倍的SNPs位点, 应用卡方检验基于测序深度对等位基因频率进行校正(Nielsen et al, 2011), 过滤单一序列标签(read)形成的多态性位点(singleton)及存在缺失数据的位点。应用PopLDdecay 3.31 (Zhang et al, 2019)进行连锁不平衡(LD)分析, 选用间隔10 kb以上的位点, 以确保获得不连锁的独立SNPs位点。通过以上筛选获得的SNPs位点用于后续的种群遗传结构分析。
1.3.3 遗传多样性分析
应用VCFtools 0.1.15 (Danecek et al, 2011)分别对海景湾个体和南湾个体的全部SNPs位点进行分析, 计算π值。
1.3.4 种群结构分析
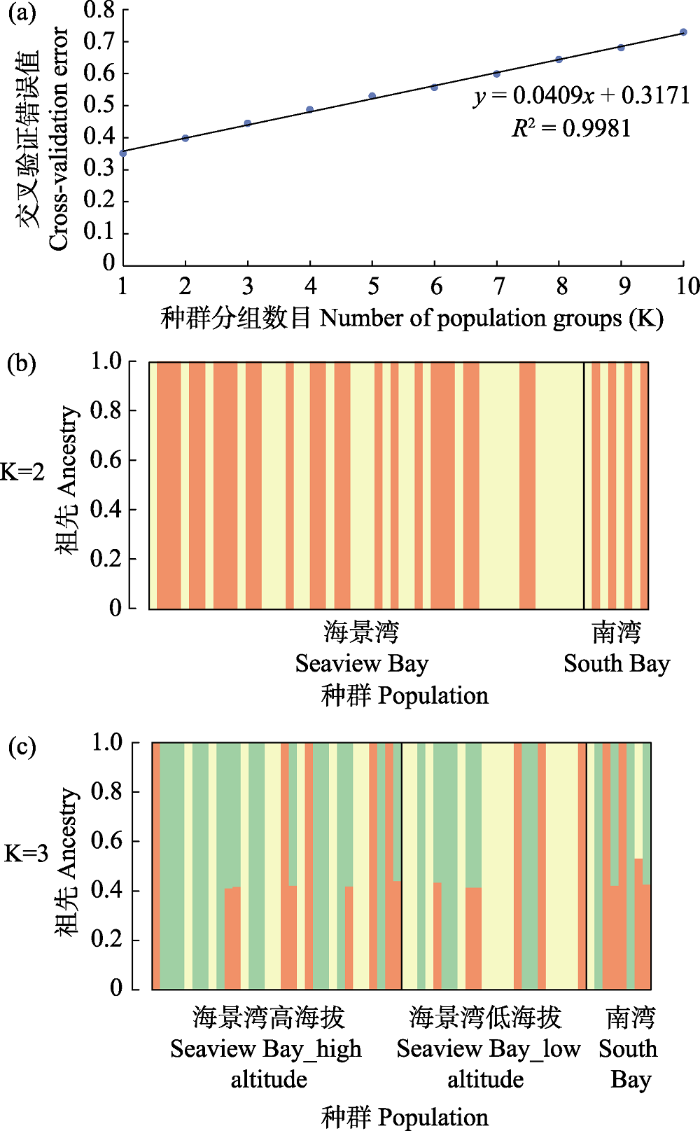
应用基于最大似然法的ADMIXTURE 1.3.0 (Alexander et al, 2009)推算种群遗传结构, K取值为1-10, 根据交叉验证(cross-validation, CV)错误值判断种群分组情况(K值), CV值最小时所对应的K值为最佳分组数目。进而在指定个体地理信息的基础上(K = 2: 海景湾区域与南湾区域; K = 3: 海景湾高海拔区域、海景湾低海拔区域和南湾区域), 进行个体的种群归属(population assignment)分析, 如果同一个地理种群的个体未形成独特的遗传单元(具有共同祖先来源), 且不同地理种群的个体具有相同的遗传来源, 则表明地理种群之间不存在显著的遗传分化。利用基于贝叶斯算法的fastSTRUCTURE 1.0 (Raj et al, 2014)分析种群遗传结构, K值设为1-10, seed值设为100, 并应用SNPRelate 1.18.1 (Zheng et al, 2012)进行主成分分析(principal component analysis, PCA), 评估种群分化程度。对亲缘关系较近的个体仅保留其中一只纳入数据分析(见1.3.5节)。
1.3.5 距离隔离效应分析
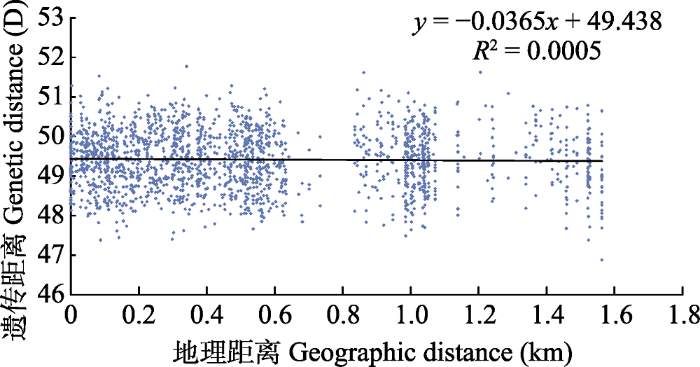
利用PGDSpider 2.1.1.5 (Lischer & Excoffier, 2012)进行数据格式转换。随机选择8,000个SNPs位点, 利用GenALEx 6.51b2 (Peakall & Smouse, 2012)计算个体间的亲缘系数, 根据Ritland’s (1996)算法得到RI值, 对亲缘关系较近(RI > 0.3)的个体保留其中一只, 纳入后续分析。结合采样点的地理坐标进行Mantel检验, 以检验是否存在伴随地理距离增加而遗传分化程度增加的距离隔离效应(isolation by distance, IBD)
2 结果
2.1 遗传多样性
测序个体中共有72只阿德利企鹅与参考基因组的比对率超过95% (海景湾高海拔区域25只、低海拔区域39只; 南湾区域8只), 平均测序深度为15.63倍。其余样品可能存在来源于环境中的DNA污染, 导致比对率较低(< 90%)。基于亲缘关系分析排除10个亲缘关系较近的个体, 应用62个个体进行后续分析。基于种群数据获取SNPs位点50,598,094个, 筛选后用于数据分析的SNPs位点共110,451个。
海景湾区域所有个体的遗传多样性(π)为0.153%, 其中高海拔为0.152%, 低海拔区域为0.153%。南湾区域所有个体的遗传多样性(π)为0.153%。
2.2 种群遗传结构
图2
图2
基于110,451个SNPs利用最大似然法构建的恩克斯堡岛阿德利企鹅的种群遗传结构。(a)交叉验证错误率分布; (b) K = 2; (c) K = 3。图中每一竖条为1个个体, 用颜色表示每个个体基因组中不同祖先所占的比例。
Fig. 2
Based on 110,451 SNPs, the population genetic structure of Adélie penguins was constructed by maximum likelihood method. (a) Distribution of cross-validation errors; (b) K = 2 and (c) K = 3, each vertical bar represents an individual, and the colours show the proportion of ancestry assigned to each of the clusters.
图3
图3
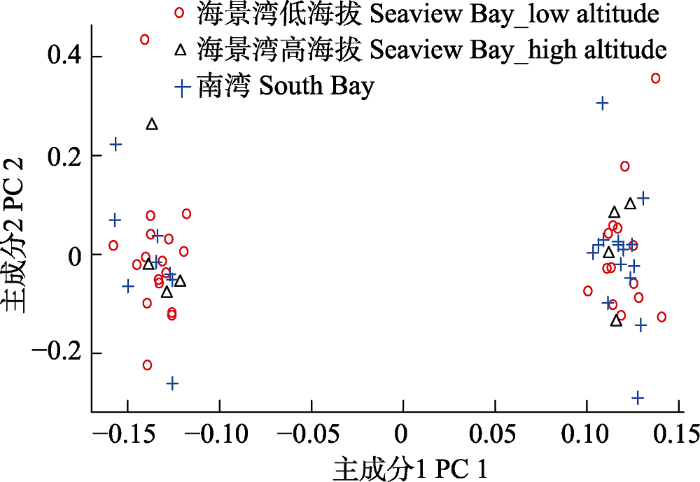
基于62只阿德利企鹅110,451个SNPs的主成分分析
Fig. 3
PCA analysis based on 62 Adélie penguins using 110,451 SNPs
2.3 Mantel检验
个体间的遗传距离与地理距离没有显著相关性(P = 0.400), 地理距离的增加并没有导致基因流受限, 不存在距离隔离效应(图4)。
图4
图4
阿德利企鹅个体间遗传距离与地理距离的Mantel检验
Fig.4
Mantel test between genetic distance and geographic distance among Adélie penguins
3 讨论
本研究对恩克斯堡岛上的阿德利企鹅进行全基因组重测序, 利用贝叶斯法、最大似然法及主成分分析等多种方法对分布于海景湾与南湾的阿德利企鹅繁殖种群进行了遗传多样性调查和种群遗传结构分析。结果表明, 海景湾与南湾附近分布的阿德利企鹅没有显著的遗传差异, 海景湾附近的海拔差异对阿德利企鹅的基因流与扩散也没有显著影响。
对于海景湾和南湾区域阿德利企鹅个体进行遗传多样性分析发现, 阿德利企鹅核苷酸多态性水平约为0.153%。Younger等(2017)对南极8个帝企鹅(Aptenodytes forsteri)繁殖地个体进行遗传多样性分析发现, 帝企鹅的核苷酸多态性水平约为0.126%, Clucas等(2016)对王企鹅(Aptenodytes patagonicus)遗传多样性分析发现, 王企鹅的核苷酸多态性水平约为0.122%。相对帝企鹅和王企鹅来说, 恩克斯堡岛的阿德利企鹅具有较高的遗传多样性, 这可能与本区域的繁殖种群持续时间较长, 且数量稳定, 保留了较高的祖先遗传多样性有关。
Shepherd等(2005)对恩克斯堡岛上48只现生阿德利企鹅的种群结构进行了研究, 他们通过9个微卫星位点等位基因频率的分析发现, 恩克斯堡岛的现生种群倾向于随机交配(P = 0.375), 可能不存在明显的种群结构。但其研究中未结合采样个体的地理信息对繁殖种群之间的遗传分化程度进行比较, 使用的少量微卫星位点数目对微地理尺度下种群结构的分析也缺乏解析力, 采样过程中没有考虑海拔是否会对阿德利企鹅群体间的基因流与扩散造成影响。本研究应用全基因组水平上的SNPs数据对种群遗传结构的分析结果更为精确, 为恩克斯堡岛阿德利企鹅繁殖群体之间不存在显著的遗传分化提供了充足的证据, 同时也支持Shepherd等(2005)通过标记观测所推测的, 本地区阿德利企鹅的归巢行为并没有阻隔海景湾和南湾区域繁殖群体间的基因交流。
本研究中主成分分析表明个体间存在一定程度的分化, 但个体间遗传分化情况与地理分布格局无关。推测可能是祖先遗传多样性的保留, 或者该地区的繁殖种群来自两个曾经隔离的避难所。基于现有数据无法确定遗传分化的来源, 有待于在更广泛采集样品的基础上, 进行演化历史的研究。
精确划分繁殖地边界对于制定精准的保护策略具有重要意义。1994年对全球阿德利企鹅进行首次普查时即提出了应当划分一个大的繁殖地或是几个小繁殖地的保护管理问题, 并建议遵循以往数目调查时的边界划分来定义阿德利企鹅繁殖地边界(Lynch & LaRue, 2016)。在本研究中, 利用全基因组水平的SNPs位点进行分析, 表明恩克斯堡岛内不同阿德利企鹅种群之间均不具有显著的遗传分化, 不同繁殖小群之间存在频繁的扩散, 基因流较强, 应属于同一个保护单元, 各繁殖小群不具有独特的保护单元地位。因此, 可以将南湾和海景湾归为同一个大繁殖地。而且, 海景湾的种群数量远远多于南湾, 应作为重点保护种群, 建议保护区划界时涵盖海景湾繁殖种群的分布范围。
致谢:
感谢第34次南极科学考察队和罗斯海新站建设队在项目执行中的大力支持, 感谢中国科学技术大学高月嵩、黑龙江测绘地理信息局朱李忠在采样和地图绘制中的帮助。
参考文献
Fast model-based estimation of ancestry in unrelated individuals
A new Holocene relative sea-level curve for Terra Nova Bay, Victoria Land, Antarctica
Holocene raised beaches at Terra Nova Bay, Victoria Land, Antarctica
Multi-modal survey of Adélie penguin mega-colonies reveals the Danger Islands as a seabird hotspot
Katabatic wind forcing of the Terra Nova Bay polynya
Comparative population genomics reveals key barriers to dispersal in Southern Ocean penguins
Dispersal in the sub-Antarctic: King penguins show remarkably little population genetic differentiation across their range
Antarctic seabird and seal monitoring studies
The variant call format and VCFtools
A 45,000 yr record of Adélie penguins and climate change in the Ross Sea, Antarctica
The Sentieon Genomics Tools—A fast and accurate solution to variant calling from next-generation sequence data
Harnessing genomics for delineating conservation units
Population genetic structure and gene flow of Adélie penguins (Pygoscelis adeliae) breeding throughout the western Antarctic Peninsula
Fast and accurate long-read alignment with Burrows-Wheeler transform
PGDSpider: An automated data conversion tool for connecting population genetics and genomics programs
Stable isotopes reveal Holocene changes in the diet of Adélie penguins in Northern Victoria Land (Ross Sea, Antarctica)
Trends in the breeding population of Adélie penguins in the Ross Sea, 1981-2012: A coincidence of climate and resource extraction effects
Landscape genetics: Combining landscape ecology and population genetics
Genotype and SNP calling from next-generation sequencing data
Identification of management units using population genetic data
GenAlEx 6.5: Genetic analysis in Excel. Population genetic software for teaching and research—an update
fastSTRUCTURE: Variational inference of population structure in large SNP data sets
Ancient DNA enables timing of the Pleistocene origin and Holocene expansion of two Adélie penguin lineages in Antarctica
Microevolution and mega-icebergs in the Antarctic
Air census of two colonies of Adélie penguins (Pygoscelis adeliae) in Ross Dependency, Antarctica
Ecological responses to recent climate change
The status and trends of Antarctic and sub-Antarctic seabirds
The challenges of detecting subtle population structure and its importance for the conservation of emperor penguins
PopLDdecay: A fast and effective tool for linkage disequilibrium decay analysis based on variant call format files
Comparative genomics reveals insights into avian genome evolution and adaptation
A high-performance computing toolset for relatedness and principal component analysis of SNP data